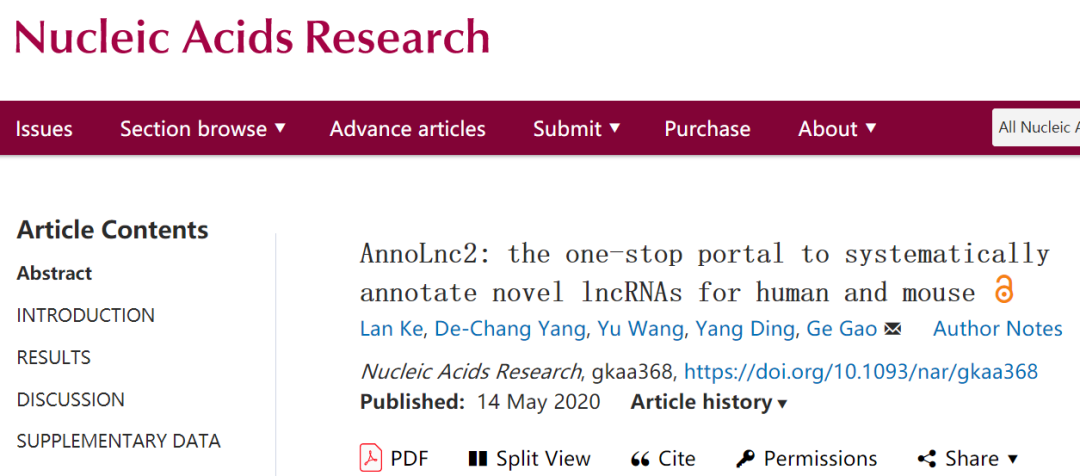

图1. AnnoLnc2框架
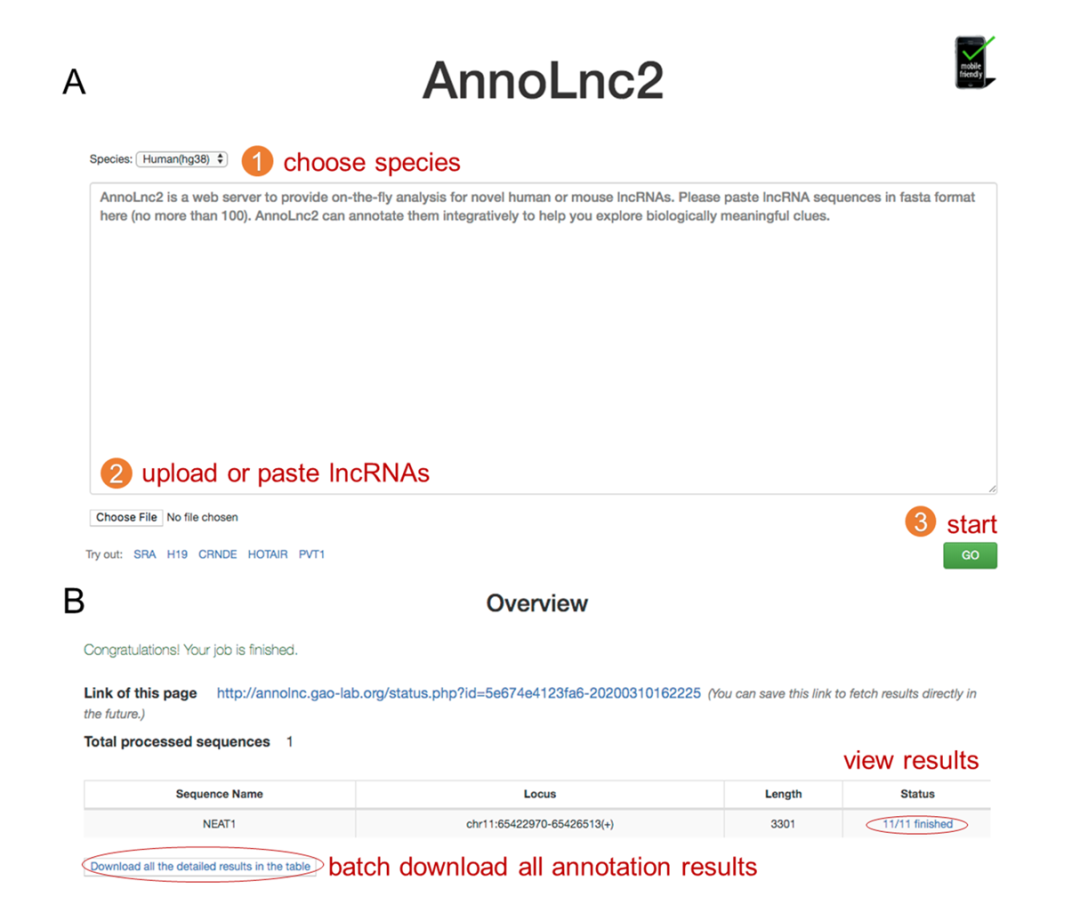
图2. AnnoLnc2 网页界面。用户可通过一个三步骤的操作运行AnnoLnc2(A),并查看详细的注释结果,以及批量下载所有的注释结果(B)
AnnoLnc2网址:http://annolnc.gao-lab.org/
原文链接:https://doi.org/10.1093/nar/gkaa368
参考文献:
1. Batista,P.J. and Chang,H.Y. (2013) Long noncoding RNAs: Cellular address codes in development and disease. Cell, 152, 1298–1307.
2. Wapinski,O. and Chang,H.Y. (2011) Long noncoding RNAs and human disease. Trends Cell Biol., 21, 354–361.
3. Faghihi,M.A., Modarresi,F., Khalil,A.M., Wood,D.E., Sahagan,B.G., Morgan,T.E., Finch,C.E., St. Laurent,G., Kenny,P.J. and Wahlestedt,C. (2008) Expression of a noncoding RNA is elevated in Alzheimer’s disease and drives rapid feed-forward regulation of β-secretase. Nat. Med., 14, 723–730.
4. Mourtada-Maarabouni,M., Pickard,M.R., Hedge,V.L., Farzaneh,F. and Williams,G.T. (2009) GAS5, a non-protein-coding RNA, controls apoptosis and is downregulated in breast cancer. Oncogene, 28, 195–208.
5. Gupta,R.A., Shah,N., Wang,K.C., Kim,J., Horlings,H.M., Wong,D.J., Tsai,M.-C., Hung,T., Argani,P., Rinn,J.L., et al. (2010) Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature, 464, 1071–1076.
6. Frankish,A., Diekhans,M., Ferreira,A.M., Johnson,R., Jungreis,I., Loveland,J., Mudge,J.M., Sisu,C., Wright,J., Armstrong,J., et al. (2019) GENCODE reference annotation for the human and mouse genomes. Nucleic Acids Res., 47, D766–D773.
7. Jiang,S., Cheng,S.-J., Ren,L.-C., Wang,Q., Kang,Y.-J., Ding,Y., Hou,M., Yang,X.-X., Lin,Y., Liang,N., et al. (2019) An expanded landscape of human long noncoding RNA. Nucleic Acids Res., 47, 7842–7856.
8. Zhou,J., Huang,Y., Ding,Y., Yuan,J., Wang,H. and Sun,H. (2018) lncFunTK: a toolkit for functional annotation of long noncoding RNAs. Bioinformatics, 34, 3415–3416.
9. Cao,Z., Pan,X., Yang,Y., Huang,Y. and Shen,H.-B. (2018) The lncLocator: a subcellular localization predictor for long non-coding RNAs based on a stacked ensemble classifier. Bioinformatics, 34, 2185–2194.
10. Su,Z.-D., Huang,Y., Zhang,Z.-Y., Zhao,Y.-W., Wang,D., Chen,W., Chou,K.-C. and Lin,H. (2018) iLoc-lncRNA: predict the subcellular location of lncRNAs by incorporating octamer composition into general PseKNC. Bioinformatics, 34, 4196–4204.
11. Li,J., Ma,W., Zeng,P., Wang,J., Geng,B., Yang,J. and Cui,Q. (2015) LncTar: a tool for predicting the RNA targets of long noncoding RNAs. Brief. Bioinform., 16, 806–12.
12. Zhang,W., Yue,X., Tang,G., Wu,W., Huang,F. and Zhang,X. (2018) SFPEL-LPI: Sequence-based feature projection ensemble learning for predicting LncRNA-protein interactions. PLOS Comput. Biol., 14, e1006616.
13. Hou,M., Tang,X., Tian,F., Shi,F., Liu,F. and Gao,G. (2016) AnnoLnc: a web server for systematically annotating novel human lncRNAs. BMC Genomics, 17, 931.
14. Ponjavic,J., Ponting,C.P. and Lunter,G. (2007) Functionality or transcriptional noise? Evidence for selection within long noncoding RNAs. Genome Res., 17, 556–65.
15. Zhang,K., Shi,Z.M., Chang,Y.N., Hu,Z.M., Qi,H.X. and Hong,W. (2014) The ways of action of long non-coding RNAs in cytoplasm and nucleus. Gene, 547, 1–9.
16. Guttman,M., Amit,I., Garber,M., French,C., Lin,M.F., Feldser,D., Huarte,M., Zuk,O., Carey,B.W., Cassady,J.P., et al. (2009) Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature, 458, 223–7.
17. Yoon,J.-H., Abdelmohsen,K. and Gorospe,M. (2014) Functional interactions among microRNAs and long noncoding RNAs. Semin. Cell Dev. Biol., 34, 9–14.
18. Castello,A., Fischer,B., Eichelbaum,K., Horos,R., Beckmann,B.M., Strein,C., Davey,N.E., Humphreys,D.T., Preiss,T., Steinmetz,L.M., et al. (2012) Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell, 149, 1393–406.



