2019年12月16日,魏文胜课题组在Genome Biology 杂志在线发表题为“PASTMUS: mapping functional elements at single amino acid resolution in human cells”的研究论文。
精准绘制蛋白质功能图谱对于研究蛋白质的作用机制十分重要。传统方法通常需要构建蛋白质的截短突变,工作量巨大且效率低下。近年来一些高通量手段比如利用错义突变进行文库筛选、利用CRISPR-Cas系统产生覆瓦式(tiling)突变并结合sgRNA富集分析等被用于相关研究,但这些方法受限于覆盖程度及位点精度,而且均不适用于隐性遗传突变类型。
魏文胜课题组开发了名为PASTMUS(PArsing fragmented DNA Sequences from CRISPR Tiling MUtagenesis Screening)的新方法(图1)。通过CRISPR-Cas介导的覆瓦式突变首先对目标基因产生覆盖度高、种类丰富的突变体文库,结合功能性筛选和对目标基因碎片化后深度测序,再利用全新的生物信息学分析方法对数据进行多重过滤,最终实现了对蛋白质功能相关位点的精确定位。该论文分别对三种毒素受体蛋白和三种抗癌药物靶标蛋白进行了功能性扫描,实现了单氨基酸精度的蛋白功能图谱绘制。除此以外,PASTMUS方法还可以广泛应用于非编码RNA、启动子、增强子等调控性元件的研究,也可以演变为蛋白进化的高效手段。
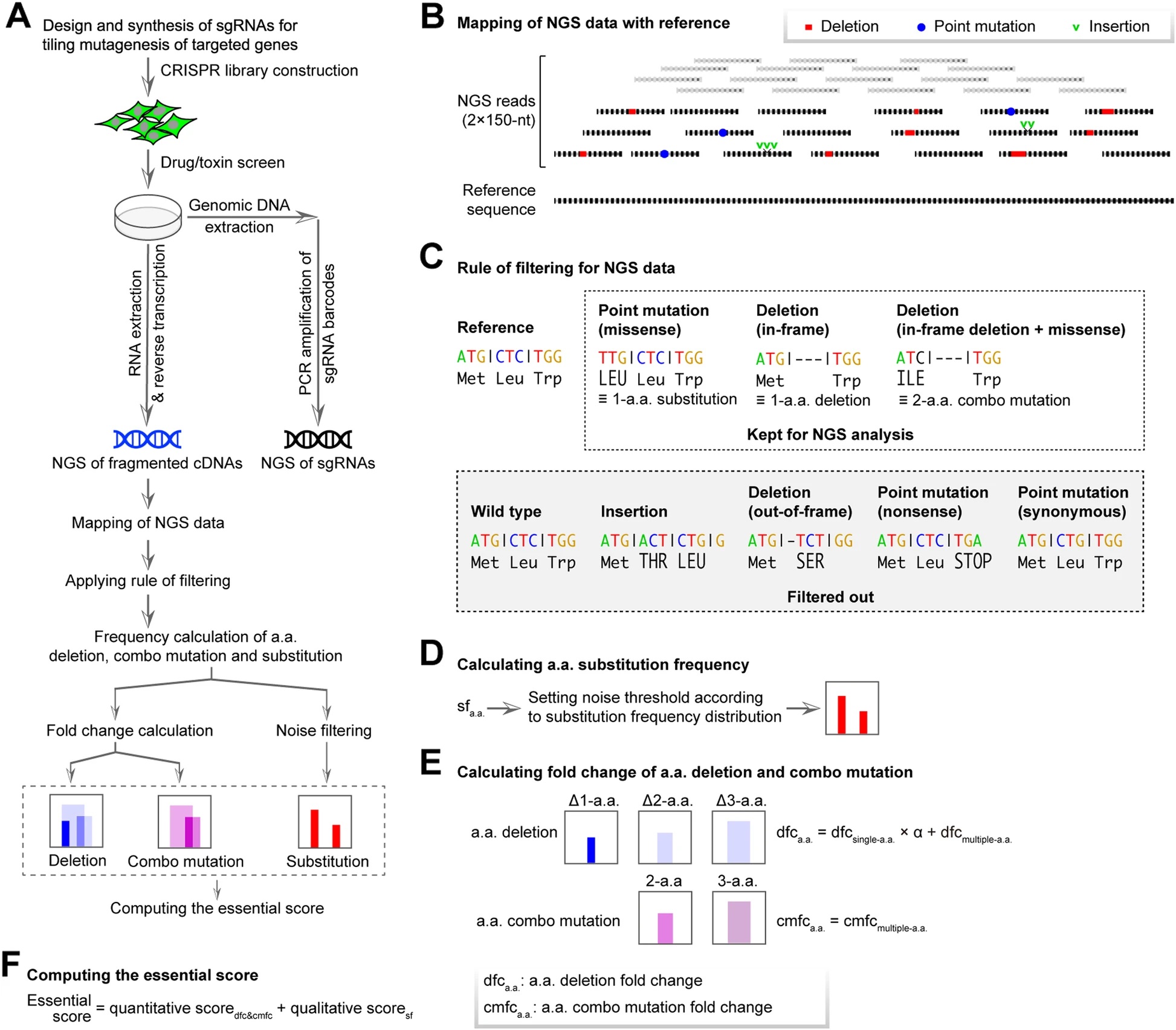
图1. PASTMUS方法筛选及生物信息学分析流程图
魏文胜课题组博士生张心怡、岳頔、博士后刘莹以及已经毕业的王轶楠博士、周悦欣博士为该论文共同第一作者。该研究项目得到了国家自然科学基金重点、面上及青年项目、北京市科委生命科学前沿创新培育项目、北京未来基因诊断高精尖创新中心、北大-清华生命科学联合中心以及传染病防治国家科技重大专项的基金支持。
文章链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-019-1897-7



