2019年10月21日,北京大学生物医学前沿创新中心(BIOPIC)、生命科学学院白凡课题组与美国亚利桑那大学姚广课题组合作在《美国国家科学院院刊》(PNAS)上发表题为“Graded regulation of cellular quiescence depth between proliferation and senescence by a lysosomal dimmer switch” 的研究论文,首次揭示了溶酶体功能在调控真核细胞静息状态深度(quiescence depth)中扮演“调节开关”的重要角色,同时也阐释了细胞静息状态加深与衰老过程分子机制上的一致性。该研究成果对于理解组织稳态、修复和再生以及机体衰老等重要生理学过程具有重要意义。
正常生理状态下,机体内的大部分细胞都不进入活跃细胞周期,而处于休眠的状态。其中,可以被生长信号刺激逆转的休眠状态被称为细胞静息,而与之形成对比的是不可逆的细胞衰老状态(senescence)。细胞静息对于机体多种生理过程至关重要,例如,干细胞稳态维持和组织的修复。另一方面,细胞静息状态的紊乱会导致诸如癌症和衰老等严重病变过程的发生。正如人的睡眠程度有深有浅那样,长期以来的研究表明细胞静息也不是一个均一的状态,而是具有不同等级的静息深度,具体可表现为细胞在接受到生长信号刺激后重新进入增殖状态(proliferation)的难易程度(例如,处于静息深度更深的细胞进入增殖状态所需要的生长信号浓度更高、诱导时间更长)。尽管以往研究对于细胞静息深度有初步的认识,然而对于调控细胞静息状态深度的分子机制以及静息和衰老过程之间的联系还一直不清楚。
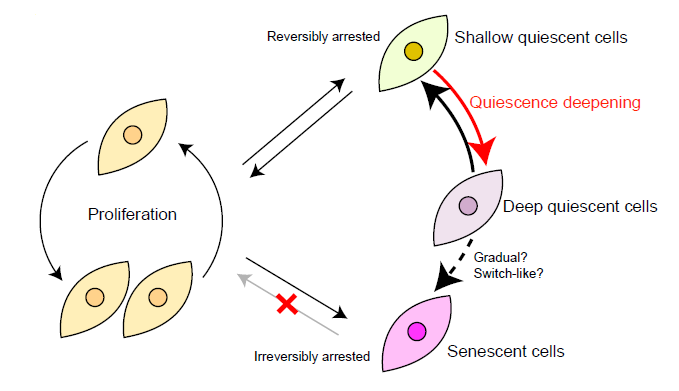
图1. 细胞的静息状态存在不同的深度
为了探究以上问题,作者团队首先利用“血清饥饿”培养的方法(0到16天)在体外诱导大鼠胚胎成纤维细胞(REF)由浅及深地达到不同深度的静息状态,并对不同深度静息状态下的细胞进行转录组测序。通过整合和分析转录组数据,作者发现在静息状态由浅及深的过程中细胞基因表达主要呈现出9种主要的模式。特别是随着静息深度的逐步加深,有两簇基因的表达量呈现逐步上升的模式。通过功能富集分析,作者发现这两簇基因都显著地富集到了溶酶体(lysosome)相关的通路上。这些溶酶体相关的基因,包括编码溶酶体相关的酶、激活蛋白、膜蛋白和离子通道蛋白的基因都随着细胞静息状态加深表达量逐步上升。同时,作者发现随着细胞静息状态变加深,细胞内溶酶体数量也随之增加。
溶酶体基因表达和数量的上升让作者猜想溶酶体的整体功能也会随之上升。然而通过对能表征溶酶体功能的自噬流(autophagy flux)进行检测,作者意外地发现随着细胞静息状态加深,溶酶体功能不仅没有增强反而逐渐降低。由此也引出了两个关键问题:1)溶酶体基因表达和数量的上升与溶酶体功能到底有什么样的关系?2)是否由于溶酶体功能的降低引起了细胞静息状态的加深?针对第一个问题,作者通过研究静息加深过程中溶酶体的生成和降解,发现随静息加深溶酶体自身降解减缓,导致破损的溶酶体数量上升而功能下降;另一方面,溶酶体基因表达的持续上调促进溶酶体生成,进而部分地补偿了溶酶体功能的降低。针对第二个问题,作者利用抑制剂抑制细胞内溶酶体功能,由此发现细胞进入了更深的静息状态,甚至不可逆的细胞衰老态。另一方面,作者利用外源表达转录因子Mitf的方式增强了细胞内溶酶体的功能,结果发现细胞的静息深度变浅。进一步研究作者发现,溶酶体功能的升高能降低细胞内活性氧自由基(ROS)的浓度防止静息状态加深。反之,溶酶体功能的降低使得细胞内ROS浓度上升进而导致静息状态加深。
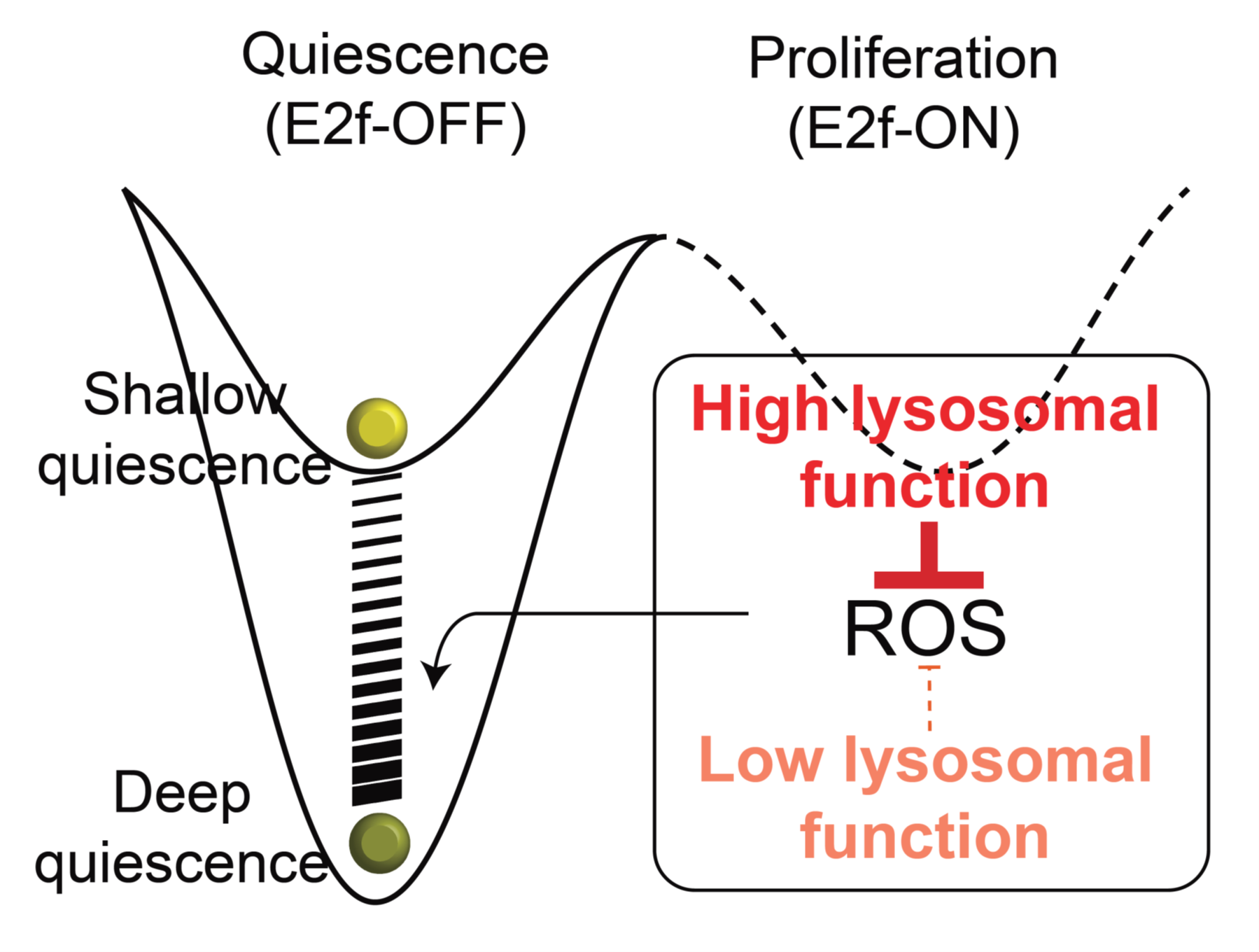
图2. 溶酶体功能在调控细胞静息状态深度中的作用
综合以上结果,作者阐明了溶酶体功能是控制细胞静息状态深度重要“调节开关”,而该“开关”的工作原理是通过调节细胞内ROS的浓度。最后,作者利用线性回归模型对转录组数据进行进一步挖掘,成功解析出能够表征细胞静息深度的一组特征基因。作者发现这组基因不仅能准确表征体内、体外细胞静息状态的深度,还能准确地预测和区分细胞衰老状态及体内多种组织的老化进程。这个结果反映了细胞深度静息状态与细胞及组织老化具有共同的分子基础,而静息状态加深是衰老过程潜在的过渡状态。
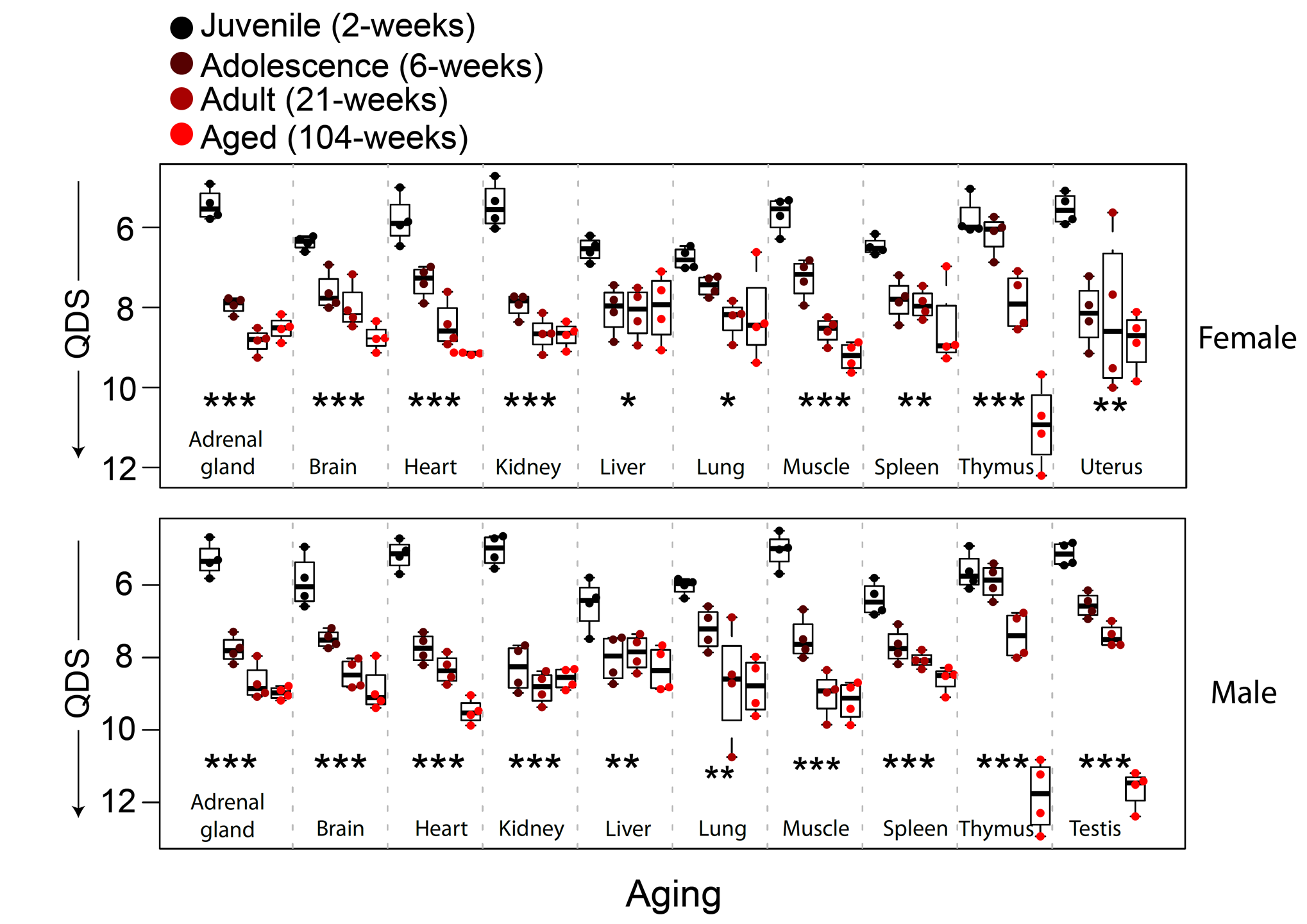
图3. 表征细胞静息状态的特征基因能准确预测和区分细胞衰老状态及体内多种组织的老化进程
北京大学白凡研究员和亚利桑那大学姚广教授为该研究论文的通讯作者。姚广课题组博士生Kotaro Fujimaki以及白凡课题组博士后李若岩为本文的共同第一作者。白凡课题组博士生陈姮玉,匹兹堡大学邢建华教授以及亚利桑那大学张灏教授也参与该研究的部分工作。该研究工作得到自然科学基金和科技部重大专项的资助。
论文链接:https://www.pnas.org/content/early/2019/10/18/1915905116



