人类基因组中97%的区域虽不编码蛋白,但仍具有不可忽视的功能,已知超过90%与疾病和性状关联的变异均位于非编码区。然而,相关变异的生物学功能与机制仍有待进一步探索。 近日,北京大学生物医学前沿创新中心(BIOPIC)、北京未来基因诊断高精尖创新中心(ICG)、北京大学生命科学学院生物信息中心(CBI)、蛋白质与植物基因研究国家重点实验室高歌课题组,通过对多个大规模实验验证表达调控变异实验产生数据的收集和整合构建了人类高质量表达调控相关非编码变异数据库REVA,并进一步评估了7个主流的非编码变异预测工具性能。论文以题为REVA as A Well-curated Database for Human Expression-modulating Variants在线发表于Genomics, Proteomics & Bioinformatics杂志。
研究团队通过对已发表文献的系统挖掘整合,提取并收录了来自18个细胞系的超过1180万个经实验验证的调控相关非编码变异,较之前发表的同类数据集增加数据量逾百倍。为理解这些非编码变异的功能与机制,课题组进一步发展了基于卷积神经网络的注释方法,对全部收录变异在转录因子结合强度、表观修饰模式、甲基化程度等多方面的影响进行了量化注释,构建了迄今为止最全面的人类表达调控相关非编码变异数据库REVA。
在此基础上,课题组进一步基于高质量变异数据集评估了7个主流的非编码变异预测工具,发现相应工具的灵敏度仍有待提升,在大规模分析中现有工具造成的假阴性是亟待注意的问题。 值得注意的是,本研究发现参与评估的工具在不同类别(细胞系/位点保守性/性状相关/疾病功能相关)的变异上都会有表现上的差异,相关现象一方面反映了非编码变异影响表达调控的复杂性,同时也提示了相应计算方法进一步改进的方向。
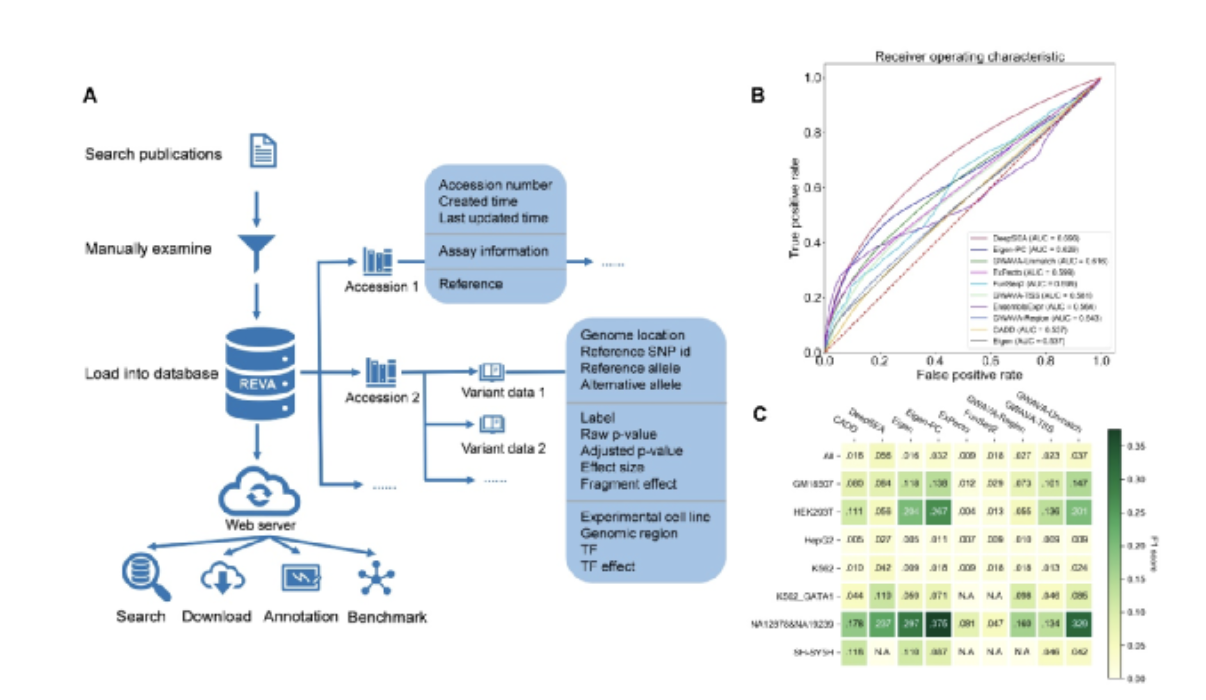
图一:REVA数据库结构概览和工具评估部分结果。A. REVA数据库整体结构的概览。B.不同工具在评估数据集上的表现。C. 不同工具在来自不同细胞系的变异上的表现。图中“All”代表不分细胞系的结果。
目前,REVA数据库已通过网络正式发布(http://reva.gao-lab.org)。用户可以通过输入染色体位置、rs id、基因名、ensembl gene id或者疾病名对变异进行快速搜索,也可以通过高级搜索页面进行自定义搜索和批量搜索。用户可以通过在线平台获取变异的基本信息、细胞系和表达信息、三维基因组信息、染色质状态、相关疾病和性状、实验相关信息和功能注释信息等,也可以下载变异的功能注释做进一步的个性化分析。同时用户也可以在线查看对现有预测工具的评估结果。
REVA数据库将持续保持更新,有相关问题可及时邮件联系reva@mail.cbi.pku.edu.cn
北京大学生命科学学院博士生王宇和史方圆为该论文的共同第一作者,高歌研究员为通讯作者,南昌大学生命科学学院硕士梁钰在数据收集上提供了大力支持。本研究工作得到了北京未来基因诊断高精尖创新中心、国家重点研发计划“精准医学专项”的支持,计算分析工作于北京大学高性能计算校级公共平台和北京大学太平洋高性能计算平台完成。
论文链接: https://www.sciencedirect.com/science/article/pii/S167202292100142X
杂志要文译荐(中文):



