哺乳动物细胞的转录调控是迄今发现的最复杂的调控系统之一,对基因的选择性表达和细胞分化等过程起主要控制作用。细胞在热休克应答中快速改变基因表达以应对热损伤,然而,基于经典蛋白质-DNA相互作用的模型并不能充分解释由急性应激触发的显著转录激活,HSF1快速并可逆地调节热休克基因转录的分子机制仍未知。
2022年3月7日,膜生物学国家重点实验室、北京大学生物医学前沿创新中心(BIOPIC)、北京大学未来技术学院国家生物医学成像科学中心孙育杰课题组在Nature Cell Biology杂志上在线发表了最新研究成果,论文题目为Reversible phase separation of HSF1 isrequired for an acute transcriptional response during heat shock。作者综合使用超分辨显微成像、体外重构和多组学等技术,建立了一个精细调节HSF1相分离的模型来探索热休克条件下转录激活的分子机制。

热休克会诱导细胞核内应激颗粒/小体(nuclear stress body,nSB)的产生,它们通常被视为热休克反应的指标。然而,研究表明,不同于酵母细胞中HSF1应激颗粒存在于伴侣基因位点,在哺乳动物细胞中的HSF1 nSB与HSP基因并不共定位【1】。相反,这些巨大的nSB在热休克期间驱动一类长的非编码RNA(卫星III转录本)的产生,这同时阻隔了转录机器,进而诱导全局转录抑制【2】。此外,啮齿动物细胞在热休克反应中并不形成nSB,但仍然可以激活HSP的表达响应热休克【3】。这些都表明广泛研究的HSF1 nSB不太可能是热休克期间发生的HSP基因剧烈转录激活的主要驱动因素。因此在nSB对HSP基因转录抑制存在的情况下,HSF1如何快速激活HSP的转录亟待研究。作者利用随机光学重建超分辨显微(STORM)技术量化了HSF1分子的空间分布。结果表明,在热休克条件下,HSF1分子在细胞核中同时形成nSB和小的凝聚体(直径~300 nm),不同于与HSP基因无重叠的nSB,这些HSF1小的凝聚体常与HSP基因位点共定位。
基于热休克期间HSF1的翻译后修饰(PTM)调控其转录活性的报道,作者通过构建一系列突变体和体外磷酸化HSF1,阐明了特定位点的PTM驱动HSF1相分离。为了进一步探究HSF1相分离对转录调控的影响,作者使用双色超分辨成像发现HSF1招募转录机器共相分离,形成转录活跃中心。同时,使用Cut&Tag和高通量测序研究了HSF1液-液相分离对其靶向染色质的影响,绘制了不同条件下HSF1的全基因组结合图谱。结果显示,相分离可以显著促进HSF1靶向染色质,而相分离缺陷的突变体结合染色质效率显著降低。最后,RNA测序与qPCR实验结果共同支持HSF1的相分离在激活分子伴侣基因表达中的关键作用。HSF1不仅在急性应激期间诱导分子伴侣表达,还在病理状态下广泛调节其他基因的表达,例如肿瘤和神经退行性疾病【4】。有趣的是,作者发现肿瘤中激活状态的HSF1不能发生相分离。而热激过程中处于相分离状态的HSF1不能激活肿瘤中HSF1的靶基因,表明细胞在急性应激和慢性过程中通过不同的机制激活靶基因的表达。近几年的研究逐步确立了相分离是激活基因表达的一种有效机制,但对于该进程如何适时终止尚缺乏研究。作者发现HSF1靶向激活表达的分子伴侣HSP70可以负向调控HSF1相分离,表现为HSP70能减弱HSF1的液-液相分离,甚至可以阻止HSF1在延长的热休克过程中发生的液固相转变,揭示了一个相分离调控基因表达的反馈机制。
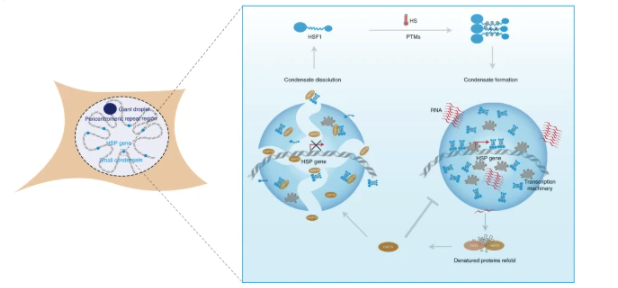
综合以上结果,该研究围绕HSF1的可诱导和可逆相分离阐明了在热休克过程中转录调控的分子机制。该机制揭示了HSF1活性的动态调节过程,即在急性应激期间有效驱动HSPs基因转录并在热休克停止后适时终止转录,维持细胞内蛋白质稳态。
北京大学生命科学学院博士研究生张宏晨和北京安贞医院助理研究员邵世鹏博士为文章第一作者,邵世鹏博士和膜生物学国家重点实验室、北京大学生物医学前沿创新中心(BIOPIC)、北京大学未来技术学院国家生物医学成像科学中心孙育杰教授为本文通讯作者。北京安贞医院曾勇教授,北京大学博士研究生王啸天、覃艺芝,中国科学技术大学项晟祺教授、博士研究生任秋楠,北京大学肖俊宇教授、王禹心博士等对本文做出了重要贡献。
论文链接:
https://www.nature.com/articles/s41556-022-00846-7
参考文献:
1.Cotto,J., Fox, S. & Morimoto, R. HSF1 granules: a novel stress-induced nuclear compartment of human cells. J Cell Sci 110, 2925-2934,doi:10.1242/jcs.110.23.2925 (1997).
2.Goenka,A. et al. Human satellite-IIInon-coding RNAs modulate heat-shock-induced transcriptional repression. J Cell Sci 129, 3541-3552, doi:10.1242/jcs.189803 (2016).
3.Denegri,M. et al. Human chromosomes 9, 12,and 15 contain the nucleation sites of stress-induced nuclear bodies. Mol Biol Cell 13, 2069-2079, doi:10.1091/mbc.01-12-0569 (2002).
4.Gomez-Pastor,R., Burchfiel, E. T. & Thiele, D. J. Regulation of heat shock transcriptionfactors and their roles in physiology and disease. Nat Rev Mol Cell Biol 19,4-19, doi:10.1038/nrm.2017.73 (2018).



