氨基酸是组成蛋白质的基本单位,其突变可以通过多种方式影响蛋白质功能,进而影响细胞生存和多种疾病的发生发展。此前研究已证明,使用碱基编辑器进行内源性氨基酸替换,并结合细胞筛选来大规模研究其功能的可行性1–3。磷酸化是细胞中常见且重要的翻译后修饰,绝大多数发生在丝氨酸、苏氨酸和酪氨酸位点上。目前已报道的大规模鉴定这三种氨基酸功能的研究主要依赖于生物信息学预测4或仅聚焦于已被检测到可发生磷酸化的位点5。考虑到蛋白组中依然存在大量潜在的磷酸化位点,且这三种氨基酸也介导了其它多种重要功能,实现在全蛋白组水平对这三种氨基酸位点的大规模功能鉴定具有重要意义。
2024年9月23日,北京大学魏文胜团队在Nature Chemical Biology在线发表题为“Functional profiling of serine, threonine and tyrosine sites”的研究论文。研究基于实验室此前开发的赖氨酸功能位点筛选策略3,利用腺嘌呤碱基编辑器对全基因组范围内的丝氨酸、苏氨酸和酪氨酸位点进行靶向编辑,并在人类视网膜色素上皮细胞系(RPE1)中进行了细胞筛选,成功鉴定出3,467个功能性氨基酸位点。这些位点的突变展示了与基因敲除类似或不同的表型,尤其是大量功能性位点位于敲除不影响细胞生长的基因上,展示了从单个氨基酸层面绘制蛋白组功能图谱的重要性(图1)。
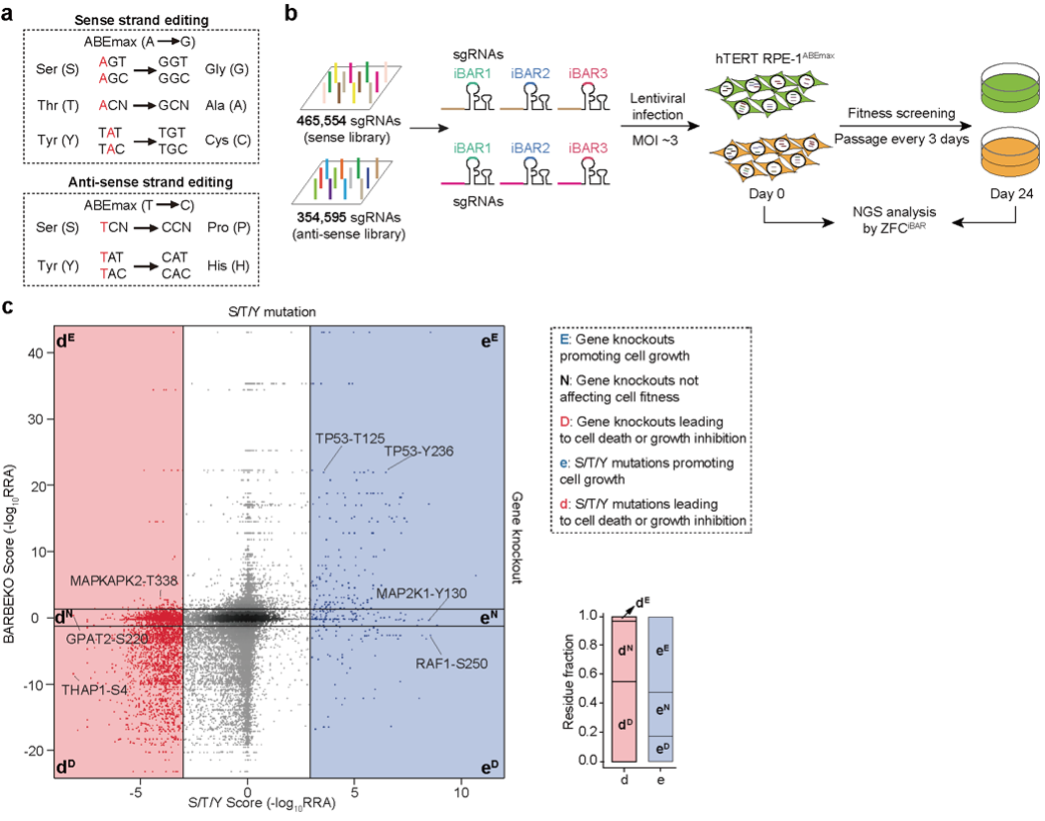
图1 蛋白质组功能性丝氨酸、苏氨酸和酪氨酸筛选示意图
接着,研究者对筛选出的功能性位点进行深入分析,特别关注磷酸化修饰。结果显示,677个筛选到的位点可能通过磷酸化修饰影响细胞生长,其中绝大多数为之前未知的功能性磷酸化位点(图2)。进一步研究表明,一些位点突变通过调节磷酸化水平导致信号通路异常,如MAPK信号通路中的MAPKAPK2T338A突变以及MAP2K1Y130H和S194P突变,影响了细胞的生长。
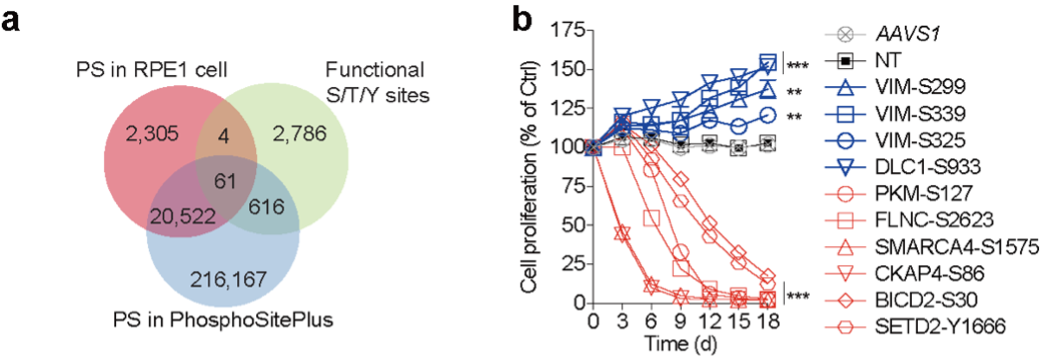
图2 筛选鉴定出大量功能性磷酸化位点
此外,研究者还分析了突变在蛋白质结构中的分布,发现丝氨酸到脯氨酸的替换广泛影响不同类型的蛋白质结构域功能,尤其是WD重复结构域中的两个高度保守的丝氨酸,暗示这些位点在维持蛋白质结构方面具有关键作用。
最后,研究发现,筛选出的促进细胞生长的突变与临床癌症患者的基因数据存在高度关联性(图3)。由于RPE1细胞系是永生化的正常细胞系,得到的309个可促进细胞生长的突变均可能为潜在的癌症驱动突变。据此猜想,研究者选择了MAP4K4-Y210C进行了体外克隆形成和小鼠体内成瘤实验,结果均证实了MAP4K4-Y210作为癌症驱动突变的潜力。
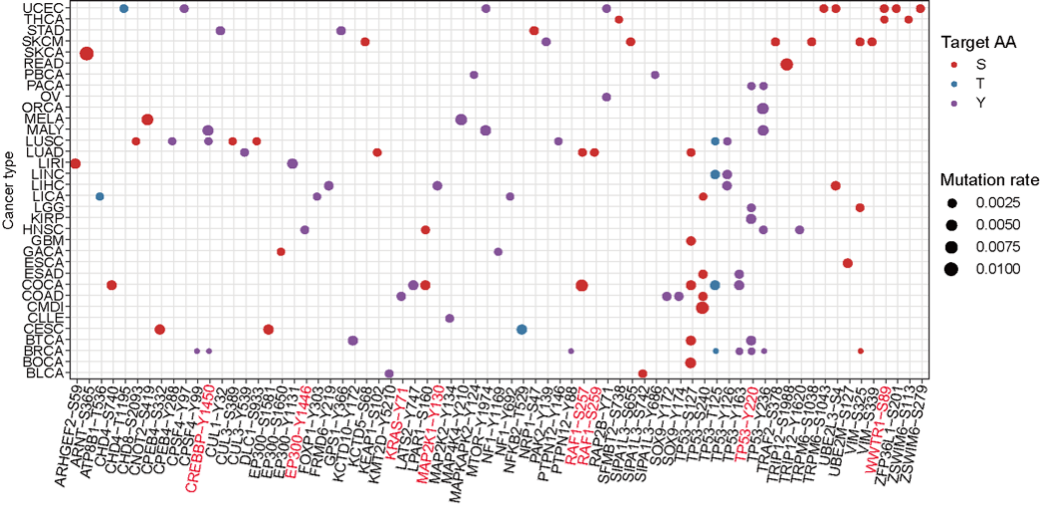
图3 与癌症临床数据相关的正向富集氨基酸位点
总体而言,该研究绘制了人类蛋白组中丝氨酸、苏氨酸和酪氨酸位点的功能图谱,揭示了这些位点与磷酸化修饰、蛋白质结构维持及癌症相关突变的密切联系,为生物学机制研究和临床应用提供了重要指导(图4)。
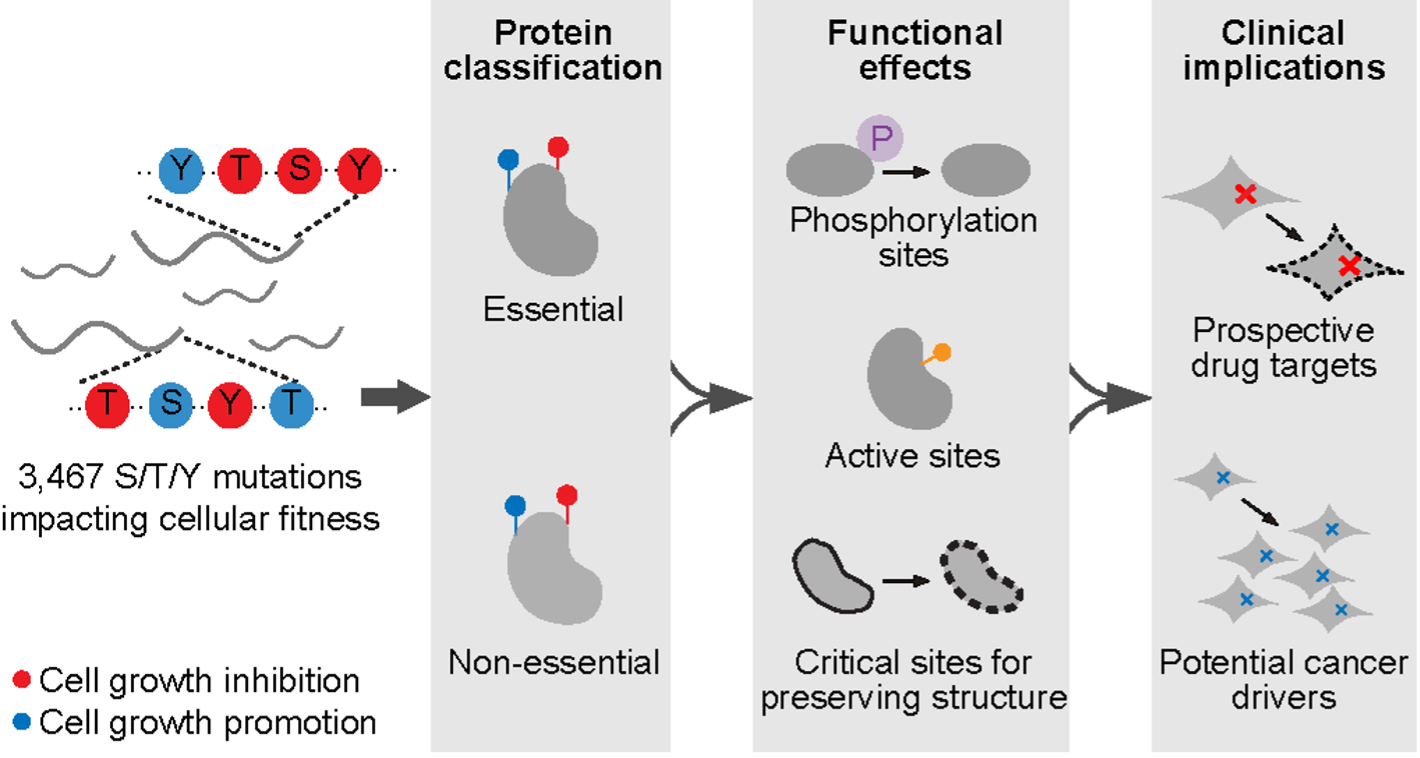
图4 研究总结示意图
本研究由北京大学/昌平实验室魏文胜团队完成,博士后李依舟、已出站的博士后徐涛及已毕业的马华峥博士为共同第一作者。研究获得了国家自然科学基金、昌平实验室和北大-清华生命科学联合中心的支持。
文章链接:https://www.nature.com/articles/s41589-024-01731-0
参考文献:
1.Cuella-Martin, R. Functional interrogation of DNA damage response variants with base editing screens.
2.Hanna, R. E. Massively parallel assessment of human variants with base editor screens.
3.Bao, Y. et al. Unbiased interrogation of functional lysine residues in human proteome. Mol. Cell83, 4614-4632.e6 (2023).
4.Ochoa, D. et al. The functional landscape of the human phosphoproteome. Nat. Biotechnol.38, 365–373 (2020).
5.Kennedy, P. H. et al. Post-translational modification-centric base editor screens to assess phosphorylation site functionality in high throughput. Nat. Methods21, 1033–1043 (2024).



