
来源: CellPress细胞科学
2021年1月23日,来自北京大学生物学前沿创新中心主任,谢晓亮教授团队在细胞出版社旗下期刊Cell上在线发表了题为Changes in genome architecture and transcriptional dynamics progress independently of sensory experience during post-natal brain development 的研究,带来了超过3000个哺乳动物脑细胞的转录组图集和三维基因组图集。这些数据可以为神经发育以及相关疾病的诊疗提供帮助。
Cell Press细胞出版社微信公众号对该论文作者团队进行了采访并对论文进行了解读,旨在与广大科研人员深入分享该研究成果以及一些未来的展望。
研究摘要
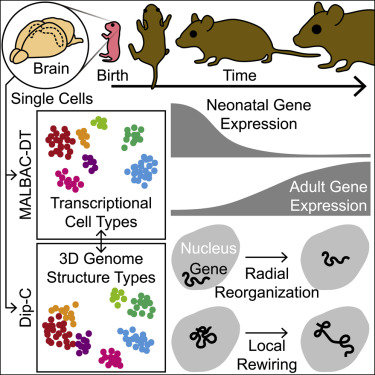
哺乳动物基因组的转录和三维(3D)结构在神经发育和发育障碍中发挥着至关重要的作用。然而,单个脑细胞的3D基因组结构仍是未知,研究界对出生后的单细胞转录组和3D基因组动态也了解很少。在此,我们利用了高分辨率的“用于数字转录组分析的多次退火环状循环扩增技术”(MALBAC-DT)和“二倍体染色质构象捕获”(Dip-C)方法,生成了正在发育的小鼠皮层和海马体的转录组(3,517个细胞)和3D基因组(3,646个细胞)图谱。在成年小鼠中,3D基因组“结构类型”描绘了所有主要细胞类型,染色质A/B区室化与基因表达之间存在很高的相关性。在发育过程中,转录组和3D基因组在出生后第一个月均发生广泛的转化。在神经元中,3D基因组大规模重新组合,这一现象与基因表达模块相关,但与感官体验无关。最后,我们探究了印迹基因的等位基因特异性结构,揭示了局部和染色体(chr)范围的差异。这些发现揭示了神经发育的未知维度。
Cell Press细胞出版社特别邀请谢晓亮教授及谭隆志博士进行了专访,请他为大家进一步详细解读。
CellPress:
请您介绍一下研究背景和初衷。
谢晓亮教授:
我们团队一直致力于单个细胞基因调控与3D基因组结构的研究。在人体的每一个细胞里,总长达2米的46条染色体具有非常独特的3D结构,能通过改变不同基因的形状来调控它们的表达。
此前,为了研究单细胞的3D基因组结构,我们团队的谭隆志博士等人于2018年开发了一项名为Dip-C的单细胞测序技术,首次获得单个人类细胞的高分辨率全基因组3D结构。直到今天,Dip-C仍是高分辨率测量单细胞3D全基因组结构的唯一方法,其分辨率比目前最先进的显微成像方法还要高两个数量级,实验操作也非常简单、成本极低。
此后的2019年,我们运用Dip-C研究了小鼠感觉神经元——鼻腔里的嗅感觉神经元、视网膜上的视杆细胞——极其特殊的3D基因组结构及其发育过程。本研究则更进一步、直接探索小鼠的神经中枢:大脑。
谭隆志博士:
基因组的结构与转录对人类健康非常重要,在大脑的发育中尤其如此。很多神经发育相关疾病——比如自闭症与精神分裂症——都与基因组3D结构与转录失调有关。然而,过去的技术无法测量单个细胞的3D基因组结构。即便在单细胞转录组层面,此前也没有哺乳动物大脑产后发育的数据。这些技术难关严重阻碍了神经发育及相关疾病的研究。本研究首次测量了哺乳动物大脑在产后发育中单细胞3D基因组结构与转录组,为神经发育研究提供了宝贵资源、为神经疾病的诊断与治疗提供了新思路。
CellPress:
能否简要介绍本研究采用的MALBAC-DT方法及其主要优势?
谢晓亮教授:
MALBAC-DT是我们团队于2020年初新研发的单细胞转录组测序方法,拥有极高的灵敏度和准确度。它的原理是基于我们团队早在2012年就已发表的高效率全基因组扩增方法MALBAC。我们曾在2015年将MALBAC应用于RNA,获得了不错的结果。作为这一系列的最新成果,MALBAC-DT在各个方面相比MALBAC RNA都有了巨大的提升,对RNA分子的扩增更高效、对表达量的测量也更准确。尤其值得一提的是,我们通过严谨的实验设计,避免了市面上绝大部分单细胞转录组测序方法都存在的RNA分子数量误测,让结果更接近真实。
谭隆志博士:
本研究中,我们首次将MALBAC-DT应用于单个细胞核,从而避免提取完整细胞时对细胞状态的不良影响。测序结果表明,MALBAC-DT果然能高效测量基因表达,检测到的RNA分子数目比以往研究小鼠大脑皮层发育的文章要多一个数量级。所以这次的转录组图谱质量非常高,更能揭示大脑发育中基因的动态表达。
CellPress:
成年小鼠和新生小鼠的神经元类型及3D基因组状态有哪些差异?
谢晓亮教授:
我们的MALBAD-DT和Dip-C数据表明,成年小鼠与新生小鼠大脑细胞在分子层面存在巨大差异。虽然新生小鼠已具有几乎所有的神经元类型,但其转录组却和对应的成年神经元不一样。一些基因只在新生神经元中表达,而另一些基因却在成年神经元中表达更高。我们把这些基因统称为新生与成年共表达模组。
谭隆志博士:
在3D基因组层面,成年小鼠的大脑神经元与新生神经元差异非常明显,看起来就像两种完全不同的细胞类型。大尺度上,我们发现基因组很多区域平时处在细胞核表面,却在神经元分化过程中大幅向细胞核内部移动;比如小鼠的7号染色体(主要对应人类19号等染色体)就几乎整条染色体内移。小尺度上,很多基因的染色质A/B区室都发生了变化,它们的增强子-启动子DNA环也随着发育改变形状。
CellPress:
本研究如何通过感觉剥夺实验证明了3D基因组转化是基因上预先确定的?
谢晓亮教授:
本研究中,我们用Dip-C方法发现大脑3D基因组结构在产后发生巨大变化。这一转化恰好发生在大脑开始接收外界感官刺激的时候,即小鼠出生后的第一个月。因此,一个非常重要的问题是:这些分子层面的变化是由外界感官刺激引起的吗?
谭隆志博士:
我们以视觉为例来研究这个问题。小鼠出生后,约12天睁开眼睛看世界。我们将出生的小鼠在黑暗中饲养,这样它们就不会受到任何视觉刺激。有意思的是,这些小鼠视觉皮层的3D基因组几乎不受影响、依然正常转化。这说明3D基因组的变化是先天决定的、不受后天影响。
CellPress:
不同功能的神经元在不同的大脑部位和/或发育阶段表现出不同的功能属性,为什么?
谢晓亮教授:
不同的大脑部位有非常不同的认识功能,其中的神经元也表现出各种独特的形态、电生理、连接特性。这些功能属性的形成都取决于神经元独有的基因表达。本研究首次得到哺乳动物大脑产后发育的单细胞转录组与3D基因组图谱,为研究神经元形成不同功能属性的分子基础提供了重要数据。
谭隆志博士:
新生儿与成年人的思维、记忆非常不一样。不同发育阶段的大脑,在认识功能和可塑性方面也有很大的区别。我们的数据表明,哪怕是在产后,不同发育阶段的神经元在基因组结构与表达上也有巨大差别。这些分子层面的变化很可能决定了大脑功能的变化,为新生与成年大脑的功能区别提供全新的解释。
CellPress:
3D基因组“结构类型”与相应的转录组类型存在怎样的关系?
谢晓亮教授:
早在2018年,我们团队就发现:仅通过单细胞3D基因组结构,就能区分人血中的主要细胞类型。因此,就像大家常说的细胞转录组类型,我们把不同的单细胞基因组结构称为“结构类型”。2019年我们研究小鼠的鼻子与眼睛,印证了这一结论。但是比起这些组织,大脑的细胞转录组类型要复杂得多,因此也更有挑战性。
谭隆志博士:
为了解决大脑细胞转录组与基因组结构的关系这一难题,我们开发了全新的数据分析方法、系统地将单细胞转录组类型与3D结构类型一一对应。我们发现,在转录组类型中特异表达的基因,其单细胞染色质A/B区室(我们称为scA/B)也会在对应的结构类型里特异地偏向A区室(即更加开放、更加真染色质)。这一独特算法让我们成功区分大脑里各种不同神经元的3D结构类型。
CellPress:
大脑神经元和OSN在基因组成和3D基因组转化方面有哪些相似之处和重要差异?该如何解释这些差异?
谢晓亮教授:
我们团队从2013年起,就一直研究OSN独特的单细胞基因表达与3D结构、并发表一系列文章。我们发现每个OSN之所以能只表达单一OR基因,是归功于它们特殊的3D基因组结构:包括OR基因在内很多区域平时处在细胞核表面,却在OSN分化过程中大幅向细胞核内部移动、多条染色体之间紧密聚集在一起,共同沉默这些OR基因。与此同时,不同染色体上的OR基因增强子也聚集起来,形成数个增强子枢纽,共同调控单一表达的OR。
谭隆志博士:
长期以来,OSN的这一3D结构一直被认为只是一个特例、没有普适性。本研究中,我们却意外发现,OSN中内移的基因组区域(包括OR基因),在成年大脑神经元里也几乎同样地移向细胞核的中心。但与OSN不同的是,不但OR和OR之间有染色体相互作用,其他类似的区域(包括免疫球蛋白在内的其他基因家族)也产生了相互作用;与此相反,OR增强子在大脑中却不再相互作用。在OSN中调控3D结构的基因(如转录因子与核纤层相关蛋白),在大脑神经元里也有不尽相同的表达特性。这说明中枢与周围神经系统的发育既有共通之处,也有略有不同的分子机制。这一结论表明,OSN因为易于取样、终生可再生,将为神经发育疾病的诊断与治疗提供宝贵材料。
CellPress:
本研究还解决了基因组学领域的哪些难题?
谢晓亮教授:
虽然在我们和其他团队的努力下,目前已出现不少单细胞3D基因组方法和数据,但相关的数据分析算法还很有限,与其他单细胞组学数据的整合方法也几乎为零。
谭隆志博士:
我们新开发的Dip-C数据分析方法,正好解决了这一困境。如上所述,我们的算法能整合分析单细胞与常规3D基因组、转录组、DNA甲基化组、开放染色质等数据,可以广泛应用于其他生物系统的研究,如癌症、器官发生、学习与记忆、衰老与神经退行等。



