来源:BioArt
哺乳动物基因组DNA全长近2 m,经过多层有序组织与折叠后分布于直径5-10 μm的细胞核中【1】。染色质高级结构对基因表达,细胞分化和胚胎发育起着至关重要的作用【2,3】。其中,基因组A/B区隔化(A/B compartments)组织形式从纤毛虫(ciliates)到人具有保守性,表明基因组内存在物种间高度保守的驱动力【4】。
回归基因组构成的本质,蛋白编码区域只有不到2%,重复序列却构成了人(Homo sapiens)和小鼠(Mus musculus)超过一半的基因组【5,6】,预示着基因组重复序列很有可能对染色质高级结构具有重要意义。LINE是人和小鼠基因组中丰度较高的一类逆转录转座原件,分别占据人和小鼠基因组的17%和19%【7】,主要包含了L1(又称LINE1,LINE-1),L2和L3等类别,基因组中有超过500,000个L1的拷贝【8】。
2020年初,清华大学医学院的沈晓骅教授团队对L1、B1/Alu等重复序列的功能做了前期探究【9,10】。对人和小鼠基因组中重复序列在的分布、对基因的表达调控以及对染色质高级结构的影响等方面积累了一些实验成果(详见BioArt报道:Cell Reports | 沈晓骅团队揭示重复序列对宿主基因组结构和功能的调控作用)。

但是,重复序列如何在调节染色质高级结构方面发挥作用?重复序列是在哪个层面上影响染色质高级结构的?其中具体的分子机制还不明确。
近日,来自清华大学的沈晓骅教授与北京大学的孙育杰教授团队合作在Cell Research 杂志发表一篇题为 Homotypic clustering of L1 and B1/Alu repeats compartmentalizes the 3D genome 的文章,对基因组重复序列如何影响细胞核区隔化提供了进一步分子依据。

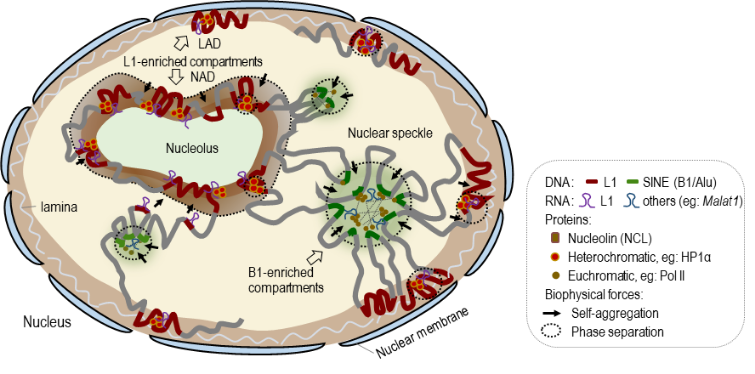
在这项研究中,研究人员首先通过系统的生物信息学分析发现,B1/Alu和L1重复序列在细胞核中倾向于与同类别DNA序列聚集分布,这种聚集特性与Hi-C推测的A/B compartments高度吻合。进一步的DNA FISH结果表明,L1聚集分布在核膜与核仁周围的异染色质区域,B1/Alu重复序列聚集分布于常染色质所在的细胞核质区域。这种差异性分布模式在mESC,NSC,NIH3T3,C2C12和HeLa细胞中高度保守,并在小鼠早期胚胎发育过程中逐渐建立,在细胞周期进程中也存在动态变化过程。
接着,研究人员利用反义核苷酸技术AMO(antisense morpholino)对mESC中L1转录本进行抑制,DNA FISH结果发现L1-rich和B1-rich的基因组区域在细胞核中的定位以及相互分离程度均受到严重影响。Hi-C数据表明AMO抑制L1 RNA会导致compartment A-A、B-B互作减弱,A-B互作增加即compartment strength [(AA+BB)/AB]降低,最终并导致染色质高级结构的改变。
最后,研究人员利用生物化学手段发现L1 DNA和RNA均能与异染色质相关蛋白HP1α发生直接相互作用,并促进HP1α蛋白在体外发生液液相分离,为理解L1如何参与调控异染色质的建立提供了可能分子依据。值得注意的是,L1与HP1α的相互作用和相分离现象并不具有序列特异性,研究人员推测两者共同的异染色质区域定位可能发挥着重要作用。
总的来说,这项研究综合利用生物信息分析、荧光标记显微成像、体外相分离体系重构等方法揭示了基因组重复序列L1和B1/Alu在compartments层面上组织染色质高级结构形成的分子机制,为深入理解小鼠和人之间高度保守的染色质区隔化结构提供了重要依据。该研究还提出了一个染色质高级结构的遗传编码机制:相同类型的重复序列相互聚集(Homotypic clustering),不同类型的重复序列相互排斥(Heterotypic segregation),这种基因组重复序列的作用模式作为先决条件奠定了染色质高级结构形成的基础;其次,重复序列以及其转录本的结合蛋白进一步调控了染色质高级结构的建立;与此同时,细胞核内无膜包被的液液相分离小体为染色质高级结构的建立提供了另外一个层面的相互作用平台。
据悉,清华大学沈晓骅教授与北京大学孙育杰教授为本文的共同通讯作者,清华大学卢金龙博士、北京大学常蕾博士、清华大学李同博士为本文的共同第一作者。清华大学尹亚飞博士(现为浙江大学独立PI)、韩雪博士和博士生王婷、战戈、张珂、陶宜冰等对本文做出了重要贡献。同时,该研究还得到了清华大学李丕龙、颉伟、那洁课题组、中科院生物物理研究所朱冰课题组和多伦多大学Miguel Ramalho-Santos课题组的大力支持。
孙育杰教授实验室的主要方向是开创、发展和应用单分子技术,包括超高分辨率单分子荧光成像技术、单分子操作及力学测量技术,以在单分子水平上研究亚细胞动态过程、精细结构与功能(Protein & Cell | 孙育杰/李程合作揭示核纤层蛋白对人源细胞中染色质高级结构及染色质运动状态的调控)。
沈晓骅教授实验室主要以多能干细胞为模型,探索非编码基因组调控染色质结构和基因表达的新机制。包括从系统和分子水平上研究非编码RNA、基因组重复序列和RNA结合蛋白,影响转录和染色质高级结构的新模式;从独特的视角来揭示干细胞多能性和细胞命运决定的普适性规律。
原文链接:https://www.nature.com/articles/s41422-020-00466-6#Sec2
参考文献
1.Annunziato, A.T., DNA Packaging: Nucleosomes and Chromatin. Nature Education 2008. 1(1):26(26).
2.Wu, J., et al., The landscape of accessible chromatin in mammalian preimplantation embryos. Nature, 2016. 534(7609): p. 652-7.
3.Wang, C., et al., Reprogramming of H3K9me3-dependent heterochromatin during mammalian embryo development. Nat Cell Biol, 2018. 20(5): p. 620-631.
4.Solovei, I., K. Thanisch, and Y. Feodorova, How to rule the nucleus: divide et impera. Curr Opin Cell Biol, 2016. 40: p. 47-59.
5.Biemont, C., A brief history of the status of transposable elements: from junk DNA to major players in evolution. Genetics, 2010. 186(4): p. 1085-93.
6.de Koning, A.P., et al., Repetitive elements may comprise over two-thirds of the human genome. PLoS Genet, 2011. 7(12)(e1002384).
7.Lu, J.Y., et al., Genomic Repeats Categorize Genes with Distinct Functions for Orchestrated Regulation. Cell Rep, 2020. 30(10): p. 3296-3311 e5.
8.Munoz-Lopez, M., et al., Study of Transposable Elements and Their Genomic Impact. Methods Mol Biol, 2016. 1400: p. 1-19.
9.Lu, J.Y., et al., L1 and B1 repeats blueprint the spatial organization of chromatin. biorxiv, 2019. 10.1101/802173.
10.Percharde, M., et al., A LINE1-Nucleolin Partnership Regulates Early Development and ESC Identity. Cell, 2018. 174(2): p. 391-405 e19.



