
由北京大学北京未来基因诊断高精尖创新中心(ICG)、生物医学前沿创新中心(BIOPIC)共同主办、为期两天的“2020单细胞组学国际研讨会(线上)”于10月16日圆满落幕。

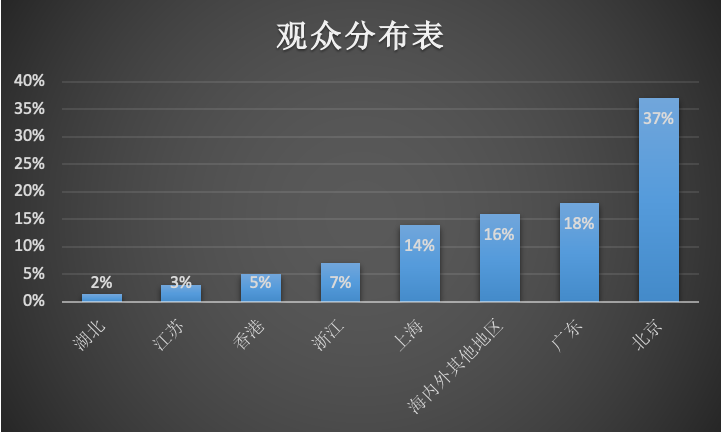
专家分布
单细胞组学国际会议系列由北京大学北京未来基因诊断高精尖创新中心、生物医学前沿创新中心的谢晓亮、汤富酬、黄岩谊三位教授共同发起,本次研讨会为第二届会议,共有21位单细胞基因组学领域的国际专家轮流做了学术报告。据统计,全球共有45830人次在线观看了此次会议。
本次研讨会得到了ILLUMINA中国、百图生科、百奥智汇及10×GENOMICS的大力支持。

研讨会伊始,谢晓亮教授作为会议主席代表致开幕辞。他首先向在线参会的广大学者及观众表示热烈欢迎。今年受新冠疫情的影响,第二届会议首次采用网上研讨的形式。在接下来的两天会期中,将有来自世界各地的21位专家为大家分享单细胞组学领域的最新研究进展。单细胞基因组学的迅猛发展令人振奋,正在走向生命科学的中心舞台。希望此次网上研讨会能惠及更多观众,进一步推动单细胞组学的传播和发展。
疾病没有国界,科研亦应如是。疫情在全球的发展轨迹正印证了国际合作的必要。唯有科学才能使人类免于新冠疫情的灾难。愿大家能在本次研讨会中积极交流,有所收获。

来自哈佛大学的David A. Weitz 教授介绍了其团队利用微流控液滴技术对微生物单细胞进行转录组和基因组测序的最新工作。微生物群体数量庞大,我们对其种类了解还缺乏详尽的了解。单细胞微生物测序是发现微生物新物种的有效工具,并且微流控液滴技术可以显著提高单细胞测序的通量和效率。Weitz教授首先介绍了利用N6随机引物代替polyT引物来进行反转录以及去除核糖体RNA这两个单细胞微流控液滴RNA测序技术的改进,从而解决了微生物RNA没有polyA,无法进行单细胞微流控液滴RNA测序这一问题。Weitz实验室还利用微流控液滴开发了Microbe-seq方法对单个微生物细胞基因组进行测序。Microbe-seq方法的亮点在于应用了两次微流控液滴融合的技术,把单个细菌细胞裂解液、下游扩增建库反应液以及带有编码引物序列的微球混合,从而实现对每个单细胞的编码。最后Weitz教授介绍了利用Microbe-seq方法的高灵敏度可以成功检测到在人体肠道菌群内新的微生物族群。

来自MIT的Blainey教授介绍了单细胞分析的两个重要工作。第一个是在单细胞基因组测序中的谱系鉴定;第二个是用于对细胞状态的检测的合并筛选新方法。准确地检测体细胞点突变是研究突变产生机制以及抗药性的基础,然而目前的技术并不成熟,突变检测常常有较高的错误,这为相关的研究带来了挑战。Blainey教授实验室创新性地开发了联合突变检测方法,将体细胞点突变的检测灵敏度和特异性提高了一个台阶,使其在谱系鉴定中有着很高的准确度。Blainey实验室开发了基于原位边合成边测序的合并筛选新方法,这种方法有着很高的准确度和检测效率。新的合并筛选方法可以实现对数以百万的人类细胞表型的的精确检测,未来Blainey实验室还计划将其应用于包括SARS-CoV-2感染的检测等多个场景中。

来自哈佛大学质谱中心的Budnik教授介绍了整合多组学技术对单细胞进行全面刻画的工作。Budnik教授首先介绍了基于质谱的单细胞蛋白组检测技术SCoPE-ME,全称是Single Cell Proteomics by Mass Spectrometry。利用SCoPE-ME,Budnik实验室能实现在单细胞层面中准确地区分出年轻和年长老鼠的胶质细胞并且鉴定出相关蛋白。SCoPE-ME目前能做到单次实验对2882个蛋白、22343个多肽的分析,并且仍有上升空间。接着Budnik教授介绍了SCREEN(Single Cell Proteome Analysis)质谱平台。SCREEN相比比SCoPE-ME在检测灵敏度上更进一步,能实现单次实验4006个蛋白,大于30000个多肽的检测效率。SCREEN的高灵敏度使得其可以被用于做特定蛋白单细胞尺度下的检测,Budnik教授最后介绍了将SCREEN用于多个代谢组学项目的研究成果。

北京大学谢晓亮教授介绍了其课题组以及合作团队所发现的新型冠状病毒(SARS-CoV-2)中和抗体的最新研究进展,其研究成果有望为新冠的治疗和预防提供强效药。利用高通量单细胞RNA和VDJ测序技术,谢晓亮领衔多个合作课题组从60个新冠康复期病人的B细胞中鉴定出20多种高效的中和抗体,其中最有效的一种是编号为BD-368-2的抗体。BD-368-2在新冠病毒感染的hACE2转基因小鼠中显示了出高效的治疗和预防功能,同时也在表现重症肺炎的仓鼠模型中也展现出良好疗效。此外,合作团队利用冷冻电镜(Cryo EM)得到了新冠病毒Spike蛋白三聚体与BD-368-2的高分辨率三维结构,揭示了BD-368-2可以通过同时占据所有三个受体结合域(RBD)来完全阻断ACE2识别。而且,BD-368-2的表位完全避免了VH3-53 / VH3-66复发性中和抗体的结合位点。BD-368-2与其复发性中和抗体配对后可以避免突变诱导的中和逃逸。谢教授表示目前该中和抗体和抗体对已在北京丹序药业有限公司的协调下与百济神州开展临床试验,有望在不久的将来为抗争新冠疫情提供有效的预防和治疗药物。

来自斯坦福大学的Garry P. Nolan介绍了细胞蛋白成像技术CODEX2和MIBI,及其在疾病治疗中的应用。CODEX2技术是通过在50-120种与细胞表面蛋白结合的抗体后面连接一段特异的核苷酸序列,之后利用带有荧光分子的互补寡核苷酸与其杂交并通过CODEX INSRUMENT进行成像,整个过程可通过反复进行多轮解链退火检测多种细胞表面蛋白。该技术发现不同组织器官具有特殊的的细胞类型排列方式,根据近邻细胞类型构建距离树有助于解释组织功能。此外通过定义肿瘤细胞和免疫抑制细胞相对与效应细胞的空间距离比值,可以实现对T细胞淋巴瘤的治疗药物的预后诊断。CORTEX对于研究新冠病毒在不同组织引发的反应也有重要作用。另一个技术MIBI是利用同位素金属离子标记抗体,之后利用质谱对细胞或组织的蛋白组成进行三维成像,该技术不仅可以测量单个细胞的蛋白质水平也同时提供个细胞形态和位置的信息,并且有望结合RNAseq实现更多的功能。

来自斯坦福大学的Steve Quake介绍了单细胞RNA测序在神经系统上的相关研究。除了已经发表的对小鼠海马体、果蝇脑中的嗅觉神经系统进行测序的工作,他们通过惊吓小鼠,令其产生害怕反应并形成长期记忆后,对小鼠的内侧前额叶皮层神经元进行单细胞RNA测序,发现有七个神经细胞子群与对照组相比存在显著的基因表达差异,同时还发现了调控差异基因表达的模体序列。此外,Steve Quake还介绍了Biohub的工作进展,目前已经完成了对小鼠18个器官的单细胞RNA测序以研究其衰老的机制,除了差异基因还发现随着年龄的增长,突变增多且克隆变大。他们也通过对鼠狐猴30个器官的RNA 测序绘制了第一份灵长类细胞图谱,并致力于人类细胞图谱的测序。

英国Wellcome Sanger Institute的Sarah Teichmann教授介绍了其团队关于人类肠道的动态转录组图谱的研究。Teichmann教授分别从上皮细胞、肠神经和肠相关淋巴组织三方面展开介绍,对来源于人类胚胎早期、中期、少儿时期和成人阶段的多个位置的肠道组织进行了高通量的单细胞RNA测序。对于上皮细胞,他们发现胚胎时期间充质细胞比上皮细胞占比更高。同时,他们鉴定出了一种新的BEST4阳性的肠上皮细胞,并表征了簇细胞的免疫感应特征及其表达的炎症性肠病(IBD)相关基因。对于肠神经细胞,他们发现了胶质和神经元的祖细胞,并找到了Hirschsprung病相关的神经细胞-平滑肌细胞的相互作用基因。对于肠相关淋巴组织,他们构建了LTi/eLTo/mLTo三细胞诱导肠相关淋巴形成的模型,并发现了在克罗恩氏病招募和保留免疫细胞的机制。Teichmann教授团队通过以上发现,结合了分子生物学特征和疾病表型,构建了全面、综合的人类肠道细胞图谱。

来自卡罗林斯卡学院的Sten Linnarsson教授的以题为《发育中大脑的分子结构》的报告介绍了其课题组围绕小鼠神经系统转录组的研究进展。其课题组将小鼠大脑和脊髓解剖为连续解剖区域后,进一步在外周感觉神经系统、肠神经系统和交感神经系统等十九个区域,采集了五十多万个细胞的转录组数据。通过分析,他们发现了七个不同的星形胶质细胞类型,以及这些受区域限制的星形胶质细胞与重要谷氨酸和甘氨酸神经递质的空间相关分布。该工作构建了详细、全面、层次分明的小鼠神经系统图谱,对小鼠神经系统各细胞类型进行了转录组注释,为理解小鼠神经系统的分子结构提供了重要资源和基础。

来自卡罗林斯卡学院的Rickard Sandberg教授的报告题目为《利用单细胞转录组解析转录动力学与加工过程》。Sandberg教授首先介绍了一种能够标记新生RNA的NACS-seq技术。这项技术通过将硫鸟苷酸(4-thiouridine,4sU)整合进新生成的RNA中,在反转录后引入C到T的突变,从而可以区分新合成的RNA鉴,实时监控细胞中RNA的动态。同时,Sandberg教授介绍了最新版本单细胞RNA测序技术,Smart-seq3。其上一版本Smart-seq2是自从2014年发表后最为广泛应用的单细胞转录组测序技术。相比于Smart-seq2,升级后的版本可通过特异性的5’端分子标签标记等位基因和异构体,并且捕获的转录本具有更高的复杂度。鉴于Smart-seq3更高的灵敏度,Sandberg教授相信其在未来的单细胞RNA测序中会有更为广泛的应用。

来自北京大学的汤富酬教授报告题目为《利用单细胞功能基因组学解析人类生殖系细胞发育》。汤富酬课题组一直以来致力于建立不同的单细胞组学研究方法,例如单细胞转录组scRNA-seq、单细胞甲基化scRRBS-seq,单细胞多组学的scCOOL-seq(甲基化和染色质可及性)和三组学scTrio-seq(转录组、甲基化和拷贝数变异)。他们利用这些技术,通过对小鼠和人类着床前胚胎、模拟着床后的人类胚胎进行单细胞分析,详细解释了受精后的胚胎早期基因表达规律和表观动态变化。除了生殖系细胞,汤富酬教授介绍了其团队对癌症发生过程的不同组学的变化规律研究。利用单细胞多组学技术,他们鉴定了结直肠癌肿瘤细胞中的亚克隆细胞群,并发现肿瘤细胞的去甲基化程度与癌旁上皮细胞原有甲基化水平呈正相关,以及肿瘤相关的成纤维细胞7号染色体有较强的拷贝数增加等现象。另外,汤富酬课题组还发现了家族性腺瘤性息肉病人的腺瘤发生、腺瘤向腺癌恶化过程中的转录组动态变化。以上发现为理解癌症发生机制提供了新思路。
来自华盛顿大学的Jay shendure教授首先介绍了高通量单细胞组学技术sci(single cell combinatorial indexing),和他们实验室基于此原理发展的多种单细胞组学技术,包括单细胞转录组技术,sci-RNA-seq。接着展示了其2019年发表的涵盖小鼠五个连续胚胎发育时期,约200万个细胞,鉴定到384个主要细胞类型的小鼠器官发生图谱,并使用monocle3算法重构了不同细胞类型的发育路径。通过整合多平台,跨多个发育时期的小鼠胚胎转录组数据数据,重构了从E3.5到E13.5的发育树,推断了每个发育分支点上的关键转录因子。另外,他还分享了其对于跨物种转录组数据整合的初步结果。最后,他报道了涵盖了15个器官(72d-129d),约400万转录组细胞和约100万染色质可及性细胞数据的关于人类胚胎发育图谱,与小鼠胚胎数据的整合,以及转录因子表达和转录因子结合基序开放性的相关性结果。

来自加州大学圣地亚哥分校的Kun Zhang教授介绍了畸胎瘤有多个组织和器官,与人类胚胎发育中的三个胚层的衍生物类似,可作为人类发育谱系研究的模型。他们利用HE染色,单细胞RNA测序等技术,对畸胎瘤的组织学和细胞类型作出了全面的鉴定,并且验证了畸胎瘤的组织结构和胚胎细胞基本类似。通过对畸胎瘤进行敲除筛选并且观察畸胎瘤的受影响区域,发现了一系列对于器官发育十分重要的基因以及转录因子,并且发现在不同的细胞谱系中转录因子有着截然不同的影响。最后他们利用分子雕刻的技术在畸胎瘤中构建Hsv-tk表达通路,筛选含有特异miRNA的细胞,从而实现对畸胎瘤的“重编程”。

来自佛罗里达州立大学的Perter Fraser教授首先介绍了他们过去几年发展的单细胞Hi-C技术和基于分子动力学模拟的染色质重构方法,在2017年他用此方法研究了染色质结构随细胞周期变化,根据相互作用的模式对细胞进行细胞周期排序。他接着讲述了最近用单细胞Hi-C研究小鼠着床前胚胎发育的工作,展示了不同发育阶段胚胎细胞周期分布的结果。通过使用谢晓亮课题组2018年发表的Dip-C算法,他重构了发育过程中二倍体细胞的结构。他首先观测了父母源染色体体积随发育阶段的变化,随后分析了染色体间相互作用模式,发现在一细胞期父母源染色体完全分离,在二细胞期父母源染色体呈现出半球分布。在四细胞时期大部分M期细胞都存在同源染色体配对的情况,分析表明可能是一种基因组自我检查机制。
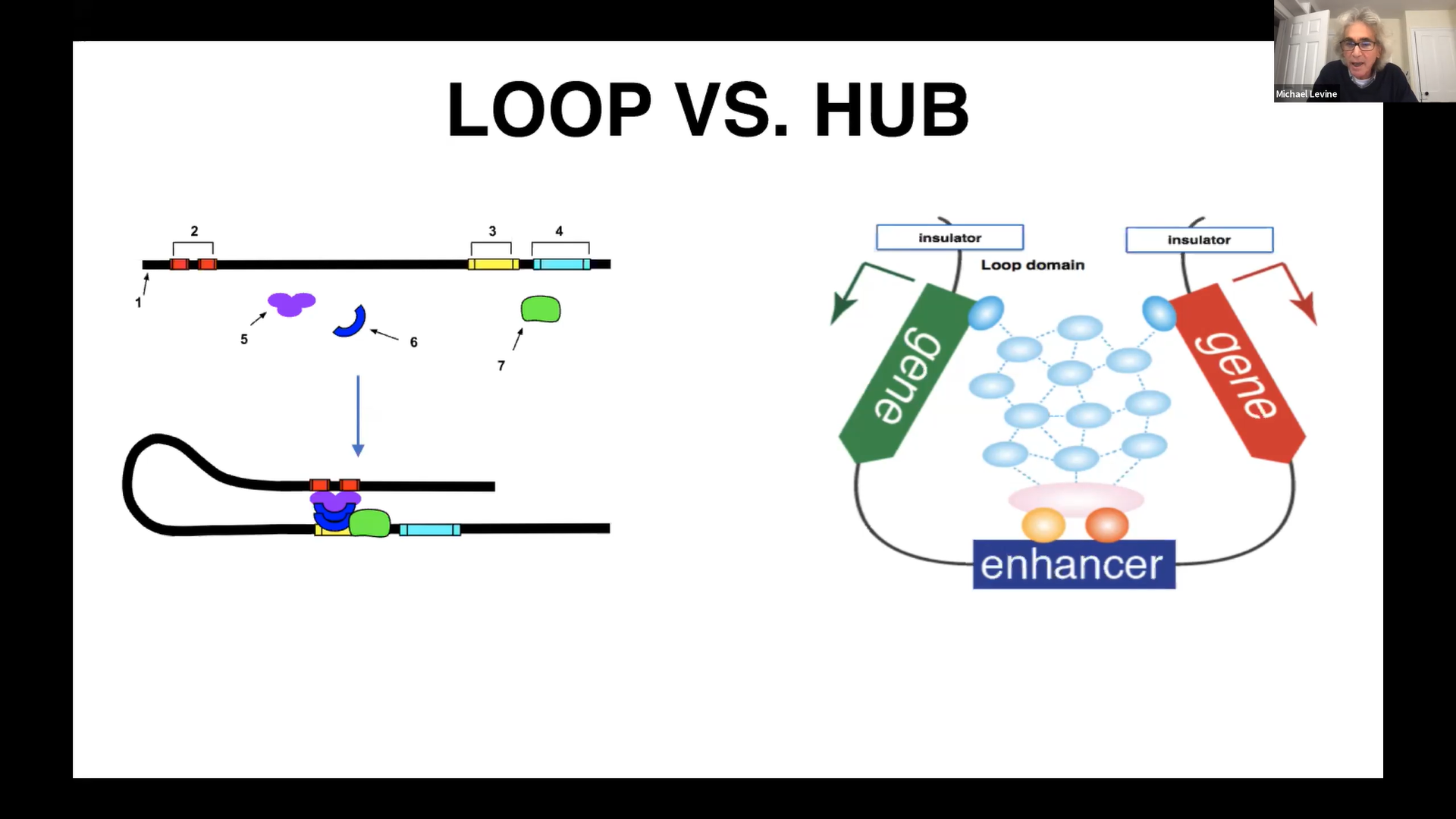
来自普林斯顿大学的Mike S.Levine教授主要介绍了其课题组两个方向的工作。一方面,他们将定量活体成像技术、基因编辑技术和Micro-C assays相结合,实现了针对活体果蝇胚胎中增强子和启动子远程互作情况的可视化。他们发现在形成转录中心的过程中,增强子发挥了核心作用。并且,Mike S.Levine教授还进一步展示了“tethering 元件”作为转录中心的关键共调节因素的证据;另一方面,他们对脊椎动物在进化中形成的新的细胞亚型充满兴趣。其课题组选择了简单的原脊椎动物”sea squirt Ciona intestinalis”作为研究对象,并基于单细胞转录组测序技术得到了Ciona高分辨率的大脑单细胞转录组数据,其中就包括该生物响应短波光(twilight)以触发变态过程的神经环路。该团队进一步发现该环路与非热带鱼类的腺泡血管有关,这是下丘脑中专门负责光周期和季节性繁殖的一种特殊结构。
来自Salk研究所的 Joseph R. Ecker在本次会议中主要介绍了他们团队利用单细胞核甲基化测序技术,绘制小鼠大脑表观基因组图谱的工作, 并介绍了多个单核测序技术,如SnmC2T-seq, SnmC-seq1/2/3, Sn-m3C-seq。基于图谱信息,他们构建了基于甲基化水平的单细胞分类法,并标注了基因特征,调控元件和转录因子。进一步,他们发现了很多有趣的现象,如:在形成既定的兴奋性和抑制性细胞类型中,相同的regulators可能会被重复使用。Joseph R.Ecker教授认为基于来自单个细胞核的多基因组数据对小鼠大脑中数百种细胞类型的表观调控水平进行注释时,该DNA甲基化图谱可作为神经元多样性和空间组织的表观遗传基础发挥巨大作用。
来自加州大学圣地亚哥分校的Bing Ren教授在报告中提出如何将GWAS发现的非编码风险位点关联到其影响的基因的问题以及其重要性。后面的报告围绕这个问题分成两个部分。第一个部分是他根据其发展的基于sci的高通量单细胞ATAC-seq技术,以及分析流程SnapATAC对小鼠大脑构建的调控元件图谱,图谱涵盖约81万细胞,根据根据转录组数据进行了注释的160个细胞亚类,并尝试通过染色质共开放鉴定到一部分远端调控元件的靶基因;第二个部分是通过他实验室最近发展的高通量联合测量组蛋白修饰位点与基因表达的单细胞技术(paired-TAG)对小鼠大脑组织进行多种组蛋白修饰测量,整合整合单细胞甲基化-染色质捕获联合测量技术,证明通过整合组蛋白修饰,染色质相互作用以及染色质可及性可以精确地预测调控元件对靶基因的影响效应。

来自北京大学的黄岩谊教授做了题为《健康人淋巴细胞中检测到低频体细胞拷贝数突变》的报告。报告分四部分进行,第一部分,黄岩谊课题组开发了一种使用Tn5转座酶快速扩增的新方法,该方法将原先需要6-15小时的扩增方法,减短至2.5小时,并通过该方法准确评估了健康人约5%有的淋巴细胞低频体细胞突变,拷贝数缺失多于拷贝数增加。同时,这种突变随机发生在全基因组上,但全染色体的拷贝数增加或缺失偏向于发生在X染色体和21号染色体上。年龄和体细胞突变关系很弱。中年女性中X染色体的单体出现率很高,推测亲本等位基因特定缺失可能和X染色体失活相关。第二部分,黄教授介绍了组里开发的3D-DPCR的技术。第三部分介绍了新开发的名为SHERRY的RNA建库测序技术。第四部分,主要介绍了北京再现的新冠疫情可能的原因是冷链食品的污染。

来自北京大学的张泽民教授做了题为《Single cell bioinformatics tools and their application in understanding tumor-infiltrating immune cells》的报告。张教授首先围绕肿瘤微环境的复杂性,对单细胞转录组测序总结了几个挑战,简单介绍了组里今年发表的生信算法:ROGUE,SciBet等。接着,张教授回顾了组里最近几年关于肿瘤微环境的研究,比较分析和归纳总结肝癌,肺癌和结直肠癌肿瘤中T细胞的分布组成和特征等,发现不同肿瘤类型中浸润T细胞的特征不同,这可能为肿瘤的临床治疗提供思路。之后,张教授也介绍了组里关于骨髓细胞,巨噬细胞等其他肿瘤微环境中免疫细胞在不同癌症类型中的特征等,期望为临床肿瘤的治疗提供新见解。
Alexander van Oudenaarden教授作报告
来自Hubrecht Institute的Alexander van Oudenaarden教授介绍了其团队利用单细胞基因组测序推测时间发育信息这一生物学问题的相关工作。利用人类结直肠癌类器官(organoids)体外培养模型,他们收集了0-25周的类器官细胞,并进行了大量细胞和单细胞测序。通过比较拷贝数的变化,以及体细胞突变和实验之初构建的慢病毒标签,Oudenaarden教授团队可以推断出类器官的具体演化过程,并观察到类器官的演化是强选择压力导致。另外,该方法可以重构致癌事件发生的先后过程,同时指明了在不同单细胞中可以独立发生相同的拷贝数变异。
免疫系统是一个复杂、动态的细胞网络,其细胞类型十分丰富。但由于历史、认知、技术限制等诸多原因,免疫细胞类型的划分一直不能统一。因此精确识别各种免疫细胞类型并阐明其分子功能是十分必要和重要的。来自Weizmann科学研究所的Ido Amit教授介绍了其实验室开发的多个单细胞基因组学测序技术,并分享了他们团队依托该技术实现的在多种生物环境下对免疫细胞类型和其可能机制的探索工作。比如利用PIC-seq技术,他们探索了分别表达配体和受体的免疫细胞间的互作机制。Ido Amit教授还介绍了单细胞基因组技术如何改变我们对从癌症到神经退行性变等各种免疫相关疾病的研究,并展望了单细胞基因组学最新和即将出现的技术和分析方法,以及它们对免疫学研究和免疫疗法未来发展的潜在影响。

来自瑞典卡罗林斯卡学院的Patrik教授首先介绍了躯体感觉神经系统,躯体感觉的不同模式,以及感觉神经元的发现过程。由于之前背根神经节中神经元种类划分的局限性,他介绍了其2017年使用单细胞RNA测序研究小鼠背根神经节的工作,此工作根据分子特征重新定义了背根神经节的神经元类型,并描述了2019年发表的关于迷走神经节的类似工作。他接着展示了其实验室结合光遗传学操控和单细胞测序鉴定躯体感觉和慢性疼痛在背根神经节上神经元投射的工作,发现感受触觉的神经元也参与了痛觉感受。最后,他分享了实验室关于非人灵长类猕猴背根神经节的单细胞转录组结果,通过比较和整合之前小鼠的数据,发现躯体感觉的神经原理非常保守,并通过整合慢性疼痛的GWAS数据,发现细胞类型特异富集的潜在致病基因。

两天的会议充实丰富,讲者们的学术报告深入浅出,听众们提出的问题也一一得到了耐心解答。研讨会在紧张有序的氛围中圆满结束。
最后,大会主席谢晓亮教授作了闭幕辞:“各位讲者的精彩分享让我们看到单细胞基因组学发展的大好未来。单细胞组学技术已经并将持续改变生物医学的研究方式,引领生命科学的发展潮流。尽管今年因为新冠疫情取消了线下活动,但线上会议的效果让我们颇感惊喜——同时在线人数高达9000人,超过45000名观众观看了本次直播。感谢会务组的辛苦付出,也感谢所有参会者的热心支持。期待明年再会!”





























