2022年10月14日,由北京未来基因诊断高精尖创新中心(ICG)、北京大学生物医学前沿创新中心(BIOPIC)共同主办的“2022单细胞组学国际研讨会(线上)”胜利闭幕。单细胞组学国际会议系列2019年由谢晓亮、黄岩谊、汤富酬三位教授在北京大学共同发起,至今已历四届。单细胞组学国际会议系列致力于全面呈现全球单细胞组学领域的最新技术开发和科研成果,不断加强我国同国际上在相关领域研究的交流合作,持续扩大我国在单细胞组学高通量测序技术领域的国际影响力。

为了让全球观众及时、快捷浏览会议相关信息,本届研讨会重磅推出官方网站平台——SCO Beijing(链接:https://www.scomics.org/),涵盖会议日程、历届讲者介绍、云展中心等板块内容,为全球网友全方位提供SCO Beijing的相关会议信息。本次研讨会聚焦单细胞基因组学、单细胞转录组学、单细胞表观基因组学及单细胞基因组三维结构、单细胞蛋白质组学、单细胞生物信息学、单细胞组学新技术、单细胞组学应用于免疫学、单细胞组学应用于癌症、单细胞组学应用于神经科学等议题。24位国际知名单细胞基因组学专家轮流展示各自实验室的最新研究进展,为国内外学者贡献了一场亮点纷呈的学术大餐。据统计,全球共有13000人次通过SCO Beijing官网在线观看此次会议,官网点赞超过52000次。
13日上午,谢晓亮教授代表会议主席团热情欢迎了在线的各位讲者嘉宾和广大观众。在开幕辞中,他表示今年是单细胞组学国际研讨会系列的第四届,相信各位观众定能看到一场最前沿、尖端的单细胞基因组学科研成果分享盛会。他提到,经讲者同意后,SCO Beijing官网后续会播出直播回放视频,感兴趣的观众可以关注后续更新。最后,他希望本次会议能延续前三届的盛况,再次贡献出一场难忘的学术盛宴,祝愿大家在会上充分交流,收获满满!
来自Stanford University的Stephen Quake教授首先介绍了单细胞转录组学的发展史。从PCR诞生开始,研究人员们就试图从细胞中获取RNA转录信息。2003年,相关学者用微阵列来检测单细胞cDNA文库,2006年测出了单细胞中成千的转录本,2009年实现了用NGS测序技术对卵母细胞和囊胚的单细胞转录组测序。现在,Stephen Quake课题组利用单细胞转录组技术,以小鼠、果蝇、白额卷尾猴和人作为研究对象,开展衰老等方面的研究。2022年,Stephen Quake课题组在《自然》杂志上发表了关于单细胞层面的异质异体共生技术的分子印记研究,通过减慢或逆转衰老过程,降低疾病风险,结果显示,细胞类型的基因表达变化与衰老进程一致。此外,他们课题组在《科学》杂志上发表了果蝇的成虫单核转录组细胞图谱相关究成果,揭示了转录因子在特化亚型方面的作用。

来自Allen Institute for Brain Science的Hongkui Zeng教授系统介绍了其团队与合作者在小鼠、非人灵长类以及人类大脑中鉴定细胞类型并研究其形态学、生理学差异的多项工作。他们利用高通量单细胞转录组测序,比较了小鼠与人类大脑中的细胞类型差异,同时揭示了大脑皮层不同区域的神经元类型的分布差异。利用Patch-seq方法,他们研究了大脑皮层不同深度层面神经元的转录组、形态学以及电生理特性的差异。Hongkui Zeng教授还讲述了BICCN(BRAIN Initiative Cell Census Network)计划,介绍其团队利用单细胞转录组测序、MERFISH等技术对小鼠全脑细胞的分类学研究成果。
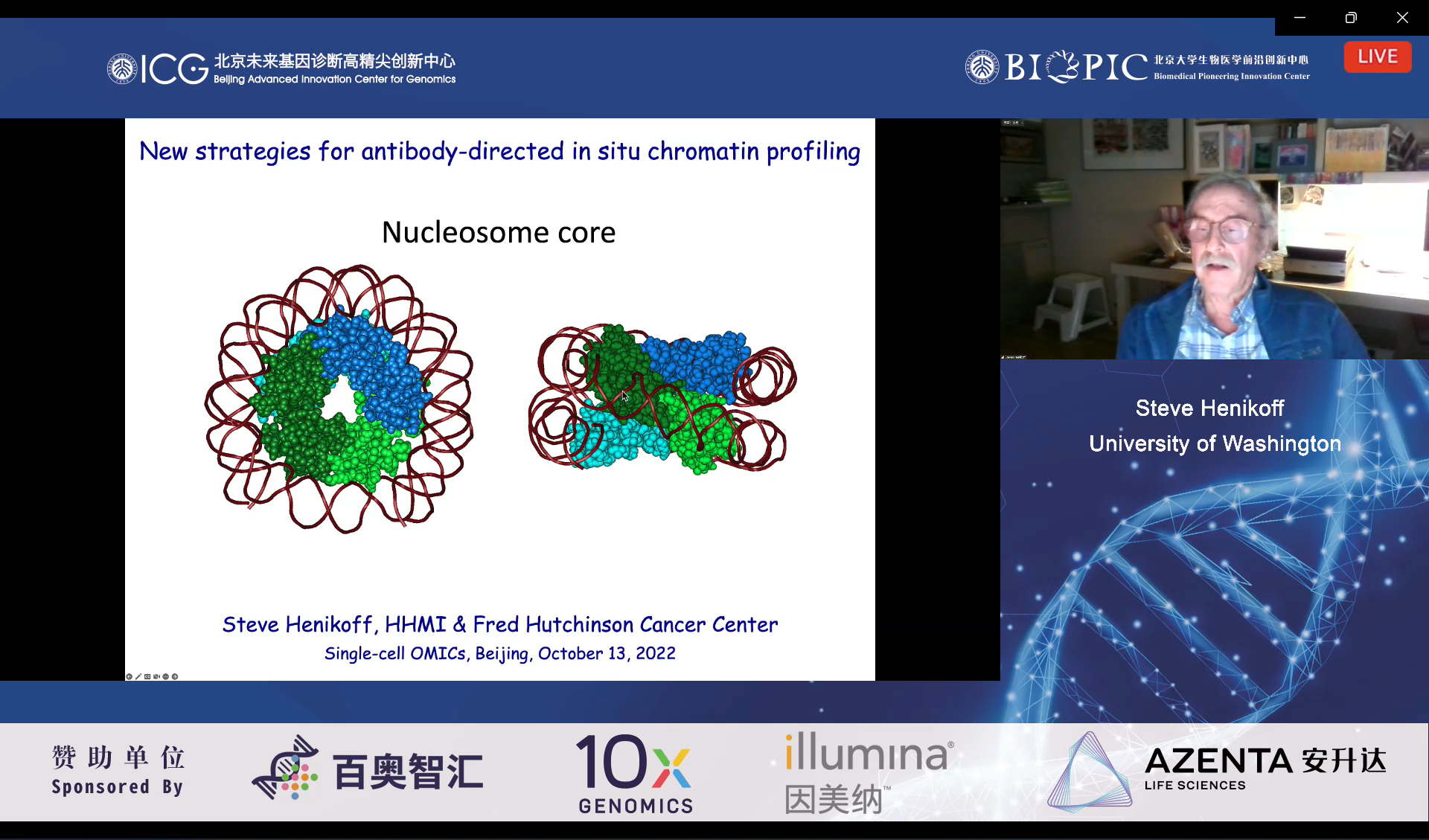
来自University of Washington的Steve Henikoff教授介绍了染色质修饰方面的最新进展。他首先回顾了染色质修饰技术的发展历程,包括基于酶标记的DamID,基于MNase切割的chEC-seq方法,以及基于Tn5标签插入技术的CUT&RUN和CUT&Tag。他的课题组在先前CUT&Tag技术基础上开发了全自动的操作平台,通过调整SDS浓度以及减少富集纯化步骤,实现了一管全程反应在一天内完成。基于该平台,他们探索了白血病中KMT2A作用H3K4me3修饰的机制。同时为了研究组蛋白多重修饰,他们课题组开发了Multi-Tag方法,实现了同时在细胞内检测多种修饰的存在。最后,Steve教授介绍了最新在《自然·方法》文章中发表的RT&Tag方法,该方法可以检测蛋白与RNA相互作用的图谱,以此研究包括果蝇RNA结合蛋白在内的各种蛋白与特定RNA的相互作用机制。

来自BIOPIC&ICG的谢晓亮教授做了题为Single-cell 3D Genome Determination with Kilobase Resolution Reveals Transcription Elongation Loops的报告。报告围绕他们鉴定到的一种新的染色质精细结构—转录延伸环进行,按鉴定、单细胞解析、特征分析、形成机制和生物学影响的顺序展开了介绍。他首先介绍了其实验室发展的高分辨率单细胞染色质三维结构研究技术—单细胞Micro-C,并用此技术研究了转录延伸环形成的动态过程。接着通过使用高分辨率染色质三维结构技术Micro-C发现拓扑异构酶抑制后下调的基因存在独特的转录延伸环—从基因转录起始位点延伸到转录终止位点,这种环通常发生在高表达的长的基因中。随后他们证明此结构是染色质结构维持复合物cohesion介导的环挤出过程依赖的,并且只有拓扑异构酶2对转录延伸环的维持是必须的。最后,他们将此结构通过影响RNA聚合酶的循环来调控基因转录动力学,有转录延伸环的基因有着更高的爆发频率。

来自University of California, San Diego的Bing Ren教授介绍了如何运用单细胞表观基因组学技术来鉴定细胞类型特异的顺式调控元件,从而确定疾病和表型特征相关的GWAS非编码变异可能影响的细胞类型和靶基因。他首先对其课题组发展的单细胞表观基因组学、多组学技术和分析算法做了综述。随后,他分享了其课题组今年发表《细胞》杂志的一项研究成果。他们通过整合其实验室生成的成人和已发表的胚胎染色质开放性图谱,鉴定到222类细胞类型中共120万个调控元件,并结合通过GWAS鉴定的疾病和特征关联的变异位点,发现了被影响的细胞类型和靶基因。在报告最后,他介绍了未发表的人类大脑染色质开放性图谱的工作,通过全面的染色质开放性图谱,他们鉴定到了神经系统疾病影响的神经和胶质细胞类型。

来自University of Washington的Jay Shendure教授讲述了其课题组高通量单细胞技术的开发和应用进展。自2015年起,Shendure课题组在不同组学中建立起基于分散-混合策略的高通量单细胞标记方法。基于该系列方法,课题组致力于绘制从线虫到人类等不同物种的全个体细胞图谱。在研究小鼠9.5到13.5天胚胎单细胞转录组的课题中,课题组整合了来自不同文献、不同技术的转录组数据,以推演细胞在不同天数下的发育路径和关键转录因子。同时,Jay Shendure教授和多个课题组合作,解析了22种基因突变对小鼠13.5天胚胎发育的影响,例如在Ttc21b敲除的小鼠中,视网膜神经的细胞类群几乎全部消失,体现了细胞比例的改变,在表达变化的分析中可检测到Sox9等转录因子在间充质干细胞发育中的动态变化。
来自Stanford University的William Greenleaf教授介绍了他在单细胞染色质可及性的方法开发与应用成果。为了研究增强子开关对基因表达的调控作用,他自2013年起开发及不断改进scATAC-seq技术,并首次在造血干细胞发育谱系中应用该技术。该数据结果表明,染色质可及性随细胞分化和基因表达的改变而改变,并且整合scATAC数据与scRNA数据可增强细胞的区分效果。近期他的课题组将该技术进一步应用于研究结直肠癌从息肉到癌肿的演变过程,发现尽管不同病人间存在异质性,单细胞染色质可及性的拟时间序列仍可以很好反映肿瘤的发展进程。此外,Greenleaf教授还致力于解释广泛的未知意义的突变对细胞表达调控的影响,他在自闭症和人类胚心研究中首次将新突变位点、染色质状态以及基因表达进行了整合分析,系统性地揭示了这些早期突变对发育相关基因调控的影响。
来自École Polytechnique Fédérale de Lausanne的Bart Deplancke教授介绍了其团队在单细胞多模态分析中开发的新工具。他首先介绍了DisCo技术。目前基于微流体的scRNA-seq仅限于具有大量细胞的样本,单个小组织样本无法得到有效处理。Disco技术将液滴技术和目标细胞的处理相结合,通过微流体阀门控制流量,借助机器视觉进行芯片的监控,实现了高效的低通量细胞处理,可应用于肠道组织的异质性研究等。随后他介绍了最近开发的Live-seq单细胞转录组分析方法。已有的单细胞组学技术需要对细胞进行分离,阻碍了对同一细胞的进一步分子和功能分析。Live-seq使用流体力显微镜,在活细胞中提取超微量RNA,同时保持细胞的生存能力,从而实现将细胞的转录组与下游分子或表型行为耦合。Live-seq可以实现scRNA-seq从端点分析到时间分析的转变,有望解决更广泛的生物学问题。

来自Karolinska Institute的Rickard Sandberg教授首先回顾了其课题组多年来开发的Smart-seq系列全长单细胞转录组测序技术。相比于目前广泛应用的基于液滴的scRNA-seq技术,Smart-seq可以捕获更多的完整转录本,对研究等位基因表达等问题具有明显优势。同时,解析等位基因的独立表达状态为研究“转录脉冲”(transcriptional bursting)的动力学特征提供了可能。Sandberg 课题组对Smart-seq系列技术进行了进一步的优化,开发了名为NASC-seq2的方法,通过4sU标记在总RNA中区分了新生RNA。通过用该方法对原代成纤维细胞的转录组测序,他们试图回答转录过程的几个基本问题:1. 转录脉冲的动力学特征是怎样的;2.等位基因之间的转录脉冲是否具有相关性;3. 是否存在相近基因转录的“共脉冲”(co-bursting)。尽管NASC-seq2 是高效灵敏的单细胞测定新生RNA的方法,初步的数据分析并不支持等位基因或者基因之间的共脉冲现象。另外,Sandberg 课题组通过优化Smart-seq实验条件和步骤,开发了更高效灵敏的单细胞RNA测序方法Smart-seq3 Express。
来自Hong Kong University of Science and Technology的Angela Ruohao Wu教授简要介绍了其课题组与Stephen Quake等课题组合作的单细胞图谱工作及重要发现。随后她主要介绍了其实验室开发的单细胞DNA-RNA多组学技术——scONE-seq。Wu教授认为单细胞多组学技术对理解癌症发生发展具有重要意义。相比之前发表的技术,scONE-seq具有更高的通量,适用于更多种类的组织(特别是冰冻组织),实验操作相对更加简单。scONE-seq在RNA层面能够精确的鉴别细胞类型特异表达基因,并通过scDASH方法去除了核糖体RNA,使之更加经济。在DNA层面,scONE-seq可以准确检测拷贝数变异。Wu 课题组利用该方法在冰冻2年的样本中鉴别出了类正常细胞的星型胶质瘤克隆细胞群,其在RNA水平上和正常星型胶质细胞较为相似,但是在DNA水平有大片段的拷贝数变化。据此,Wu 课题组首先实现了在胶质母细胞瘤中将基因型与表型联合分析,之后会在这一方面进行进一步的探索。

来自浙江大学的郭国骥教授分享了其课题组在单细胞水平对细胞研究的重大进展。是否能根据基因型推断表型?是否可以实现从描述性研究到预测性研究?他的课题组致力于用单细胞技术,阐明细胞类型的数量及调节机制,以标准化细胞的分子细胞生物学特征。他们开发了微孔测序技术,该技术具有交叉污染率低、通量高的特点。运用这项技术,他们研究了人类、小鼠、果蝇、斑马鱼、非洲爪蟾蜍、欧洲红蚯蚓等物种的细胞组成谱,发表了数篇论文。此外,他的课题组2020年在《自然》杂志上报告了基质细胞新亚型的发现。他们还发现,在小鼠等五个物种的一生中,线粒体的代谢是随着衰老持续下调的。为了研究调控细胞类型的因素,他们还研究控制细胞谱系命运的决定因素,以及解读从基因到细胞轨迹的一个常用深度学习模型。

来自Baylor College of Medicine的Chenghang Zong教授主要介绍了其在全长RNA测序领域最新的未发表成果。Zong教授团队在MATQ-seq的基础上,结合SMART-seq方法开发了名为SMART-MATQ的新方法。基于该方法,团队探索了HEK细胞系细胞周期的细胞表达谱差别。全长RNA有助于检测细胞内含子及外显子的分布,因此适合于RNA velocity的分析。基于拟时间谱系分析,可使用TradeSeq算法算出各阶段细胞周期的特征表达基因。该研究发现了三类差异细胞周期基因,一类是成熟RNA差异,表示可能在转录后调控;一类是未成熟RNA差异,表示在转录层面有差异;第三类是两者兼有。其中,第一类基因备受关注,其富集在染色体结合和微管组装的过程中;第二类基因则分布广泛,在mRNA稳定性、DNA修复及RNA代谢合成通路中各有模组。一些重要的RNA结合蛋白可能在这个过程中起到重要作用。

来自California Institute of Technology的Long Cai教授介绍了当今空间组学技术中依然还存在的一些问题,例如空间分辨率会随细胞类型而发生变化,以及目前荧光原位杂交技术的荧光数量还比较有限等。基于这些问题,Long Cai教授提出了新的技术seqFISH+,这种技术通过增加每轮成像编码数量以及荧光成像通道,大大提高了编码容量,从而实现超分辨的上万种基因的超多元成像,同时可以检测转录本在亚细胞水平的分布以及临近细胞之间的位置互作,并且能检测不同的细胞类型,这对于构建空间细胞图谱提供了非常有力的工具。最后,Long Cai教授介绍了MEMOIR,一种读取细胞的历史和谱系图的方法,能够用来重建细胞谱系树,帮助我们进一步理解发育和细胞分化。

来自 Yale University的Rong Fan教授首先回顾了空间组学的发展历史,他说到当前空间组学在空间转录组以外的其他领域发展空间还很广阔。他介绍了Spatial-ATAC-seq技术,该技术利用空间条形码技术,通过Tn5转座等技术进一步对染色质可及性在整个基因组尺度进行了空间分辨测序,与多细胞ATAC-seq有很高的契合性。通过Spatial-ATAC-seq,Fan课题组成功实现了对小鼠胚胎细胞类型的鉴定以及人体扁桃体B淋巴细胞在空间上的动态变化的检测。最后,Rong Fan教授介绍了spatial-CUT&Tag技术,实现了在空间水平检测表观遗传修饰。通过这样的技术,成功构建了小鼠胚胎的空间组蛋白修饰图谱,检测到了多种器官特异性细胞类型,这一系列工作为空间组学提供了新的视角和见解。
来自 University of Pennsylvania的 Ning Jenny Jiang教授介绍了她们团队开发的TetTCR-SeqHD技术及其应用。抗原特异性对于T细胞的功能至关重要,但是T细胞对应的抗原特异性信息在现有的单细胞分析过程中经常是缺失的。Jiang教授团队开发的TetTCR-SeqHD方法能够在单细胞水平上同时检测T细胞的抗原特异性,T细胞受体(TCR)序列,基因表达以及细胞表面蛋白表达水平,并且该方法检测抗原特异性的精度超过98%。基于TetTCR-SeqHD方法, Jiang教授团队分析了一型糖尿病患者体内CD8 T细胞的特征。与健康对照组相比,糖尿病患者体内显著富集能够识别一型糖尿病相关抗原的CD8 T细胞。此外,她的团队也观察到了免疫交叉反应(cross-reactivity)的现象,即一些与糖尿病抗原相关的CD8 T细胞克隆也能够识别特定的微生物抗原。TetTCR-SeqHD这一技术的出现会进一步加快对于T细胞的了解。
来自BIOPIC&ICG的黄岩谊教授介绍了其团队最新的两个基于测序技术开发的工作:基于荧光发生的新测序方法Bit-seq以及基于高效稀释编码的原位测序方法SPRINTseq。测序中一个重大的挑战是测序轮数增多伴随而来的失相问题,黄教授团队通过简并测序(M:A/C,K:G/T)得到简并的二值化DNA序列,并通过比对基因组得到原始的序列信息,由于每一种简并碱基在一轮反应中前进的进度不同,通过匹配可以进行失相的校正,同时该测序方法有较高的测序效率。在第二个工作中,黄教授的团队利用开发了一套抗拥挤的原位测序的技术SPRINTseq,通过设计一套高度正交的条形码序列,可以进行信号的纠错和去重叠;此外团队利用多个层次的信号稀释,最终达到了10%的稀释比例。借助于改进的实验流程和高效的SBS原位测序,在15小时内完成了小鼠全脑切片样品中目标基因的空间转录组的检测,得到了113,625个细胞108个基因的36,414,120个转录本的亚微米级分辨率的空间信息。最后,他还介绍了通过SPRINTseq分析得到的疾病中细胞及转录本亚细胞分布的变化,及由此带来的重要启发。
来自BIOPIC&ICG的汤富酬教授报告题目为Single cell omics sequencing technologies: the next generation。汤教授课题组早期建立了多种单细胞测序技术。他介绍了利用单分子测序平台对单细胞测序技术进行的革新。不同于二代测序,下一代单细胞测序技术以突破性的读长著称,从而在单细胞转录组、基因组和表观组测序上表现出了一系列优势。对单细胞转录组,长读长测序鉴定出了更多的可变剪切转录本,高效地鉴定了单个细胞表达的T细胞受体基因,区分了假基因和母基因的表达。对单细胞基因组,建立读长中位数在6 Kb的文库使得结构变异的检测变得准确而全面,也使得利用少量单细胞组装基因组序列成为了可能。对于表观组,汤教授在染色质可及性领域率先进行了探索,利用长读段捕获Tn5酶可及性染色质位点,并且在此基础上分析了纯合位点的等位基因特异性的染色质可及性,揭示了共开放现象,同时检测了基因组结构变异。总而言之,汤教授课题组通过开发下一代的长读长单细胞测序技术,迈上了探索单细胞组学“暗物质”的征程。

来自BIOPIC&ICG的张泽民教授分享了免疫治疗过程中肿瘤微环境的动态变化研究。肿瘤微环境中的复杂性,取决于其组成、细胞命运、特殊功能、细胞间相互作用、空间结构、时间动态、临床相关性和异质性。他的课题组发现,肿瘤中的LAMP3+树突状细胞是成熟的树突状细胞,具有向肝淋巴结迁移的潜在能力。他们还发现,CXCL13是响应肿瘤的T细胞上的标志物,通过对肺癌、乳腺癌等的研究,他们提出了用CXCL13的表达水平,识别免疫检查点阻断(ICB)治疗前后肿瘤微环境中肿瘤抗原特异T细胞的方法,鉴定了肿瘤特异CD8T细胞的不同分化状态及其特征,发现ICB治疗可以持续诱导CXCL13+CD8T细胞的增多,从多癌种水平揭示了ICB在细胞层面上的作用机制。

来自北京师范大学的王晓群教授介绍了他们用空间多组学研究人类小脑发育的最新工作。借助计算整合的空间染色质可及性和空间转录组,他们绘制了小脑发育中细胞命运决定的转录因子及其可及性的图谱,并发现小脑中不仅不同位置的祖细胞表现出差异的基因表达谱,发育中的神经元,如浦肯野细胞和颗粒细胞,也显示出时空特异性的分子特征。此外,通过跨物种分析,他们发现了只在人类中出现的独特细胞亚型,以及在人的浦肯野细胞中特异表达调控其分化和迁徙的因子——RORB。最后,他们发现已被报道的人类特异性基因ARHGAP11B不仅可以促进大脑皮层的扩张和折叠,在人小脑的菱唇祖细胞中也高度表达。而在小鼠的小脑中表达ARHGAP11B会促进颗粒细胞和小脑皮层扩张的增加,以及更复杂的折叠结构。

来自Hubrecht Institute的Alexander van Oudenaarden教授介绍了利用单细胞新生DNA测序揭示了基因组复制的加速度相关研究进展。通过分选合成期细胞并且标记新生DNA进行单细胞测序,这项工作检测到的复制叉与此前基于拷贝数鉴定的相吻合。为了得到复制叉移动的速度,研究者利用两次时间间隔可控的EdU给药脉冲向新生DNA引入时间信息,从而根据复制叉在固定时间内的移动距离,计算出了复制叉的移动速度。利用拟时间分析法拟合单细胞的合成期完成度,这项研究发现随着合成期接近尾声,复制速度不断增加,意味着DNA复制叉的移动存在加速度。另一方面,这项研究发现转录活动会造成DNA损伤,从而减缓DNA复制的速度。当敲除DNA损伤修复基因XRCC1之后,由于DNA损伤的累积,DNA复制速度将会减慢。这时,如果使用抑制剂阻断PARP释放的DNA损伤警报,细胞则会忽略DNA损伤继续复制,使得复制速度在一定程度上恢复。

来自Harvard University的David Weitz教授介绍了其团队利用Mcs RNA-seq和Microbe-seq这两种技术分别从转录组和基因组方面对微生物群落进行研究的最新进展。在此之前,宏基因组因灵敏度有限而无法揭示菌株之间的差异。单细胞微生物测序利用微流控液滴技术显著提高了单细菌测序的通量和效率。Weitz教授首先介绍了Mcs RNA-seq技术的两点优势:1)利用N6随机引物代替polyT引物来进行反转录;2)基于Cas9的rRNA去除技术。接着Weitz 教授团队将Mcs RNA-seq应用于环丙沙星处理的大肠杆菌,揭示其在不同时间处理过程中基因的变化。然后Weitz教授介绍了Microbe-seq方法,其亮点在于应用了微流控液滴融合技术的四部分装置和生信分析流程。该装置的各部分先后负责单细菌分离、扩增、打断和添加编码引物。对于生信分析流程,Weitz教授介绍了其先组装区分物种再利用SNV区分菌株的原理。最后Weitz教授介绍了他们利用Microbe-seq方法对人体肠道菌群的研究,其中包括他们发现从中发现的新物种,微生物群落中的水平基因转移网络和噬菌体-宿主互作。
来自Weizmann Institute的Amos Tanay教授介绍了他在转录组细胞图谱研究的进展。Tanay课题组长期关注发育过程中单细胞图谱的谱系发育情况,传统的分析包含二维的降维聚类图谱,寻找特征基因以及定量统计RNA的分布。为了更好观察细胞状态并体现生物意义,Tanay课题组开发了MCProj方法来投射新生单细胞数据到已有的细胞图谱上;相比于其他方法,该方法拥有较好的比对效率,更能发现新的细胞类群。自2019年以来,课题组在小鼠原肠胚单细胞转录组数据上进行了广泛深入研究,利用metacell拟合的拟时间序列与真实取样时间高度吻合,同时发现了不同细胞类型在发育谱中的不同发展模式。在此基础上,课题组结合兔原肠胚实验数据,基于同源基因的metacell投射方法,他们发现兔和鼠在原肠胚时期高度一致,符合早前的发育进化“漏斗”模型。该研究仍在进行之中,将为不同物种和实验中的数据整合分析提供更有力的工具。

来自BIOPIC&ICG的高歌研究员介绍了使用生成式深度神经网络模型整合单细胞多组学数据,并推断基因表达调控网络的计算方法。单细胞测序数据具有高噪声、信息量丰富、数据规模大等特点,要求计算模型具有高稳定性、可解释性且容易扩展到大数据集。他们课题组发展了GLUE方法用于多组学数据整合分析,利用生物学知识构建指导图,然后采用深度生成模型在多组学数据集上训练,得到不同组学数据在细胞类型的共同表示,在scRNA-seq+scATAC-seq的整合任务上取得最优效果。GLUE可以用于推断基因表达调控关系,并且其运行时间随数据规模的增长是亚线性的,具有很好的适用性。他们还在GLUE的基础上进一步开发了充分利用可能存在的部分细胞配对组学数据的CLUE模型,以及推广到空间组学整合分析的SLAT模型,在多项整合分析任务上取得了比Seurat等现有方法更好的结果。

来自BIOPIC的曹云龙副研究员作了题为High-throughput bisulfite-free DNA methylation and hydroxymethylation sequencing at single-cell level的报告,介绍了团队开发的基于酶学转化的单细胞DNA甲基化与羟甲基化测序技术。该方法不依赖重亚硫酸盐反应的特点极大地减少了DNA损伤,提高了基因组覆盖度。他们利用该技术分析了小鼠早期胚胎发育过程中的甲基化与羟甲基化动态变化,同时展示了亲本差异化的去甲基化模式。报告还介绍了在此前技术基础上开发的高通量、低成本的单细胞DNA甲基化与羟甲基化测序方法,并表示他的团队正在利用该方法开展针对哺乳动物大脑的甲基化与羟甲基化图谱研究工作。
两天的会议精彩纷呈,学者们的学术报告深入浅出,既分享了单细胞领域的最新科研成果,又展示了单细胞领域的技术新突破,参会观众踊跃提出的问题得到了完美解答。研讨会在紧张有序、热烈活跃的氛围中胜利闭幕。
本次研讨会得到百奥智汇、10x Genomics、因美纳、安升达的大力支持。



